您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章主要介紹“怎么使用CNVnator進行CNV檢測”,在日常操作中,相信很多人在怎么使用CNVnator進行CNV檢測問題上存在疑惑,小編查閱了各式資料,整理出簡單好用的操作方法,希望對大家解答”怎么使用CNVnator進行CNV檢測”的疑惑有所幫助!接下來,請跟著小編一起來學習吧!
CNVnator是一款CNV檢測軟件,基于Read-Depth的分析策略,通過對全基因組測序數據進行分析來預測CNV, 源代碼保存在github上,網址如下
https://github.com/abyzovlab/CNVnator
這個軟件的安裝比較復雜,我這里直接使用別人裝好的docker鏡像進行處理,這也是docker的方便之處,直接從源中下載別人已經裝好的cnvnator的鏡像,代碼如下
docker pull diploid/cnvnator
具體的分析步驟如下
CNVnator中依賴ROOT這個軟件包,這個軟件包是專門針對大數據的處理進行開發的,提供了統計分析,可視化,數據存儲等一系列功能,基于這個體系可以極大的加快運行速度,所以將原始的信息都存在root文件中,便于分析。
這一步將比對的信息存儲到后綴為root的文件中,代碼如下
cnvnator -root file.root -tree chr1.bam -chrom 1
-tree參數指定輸入的bam文件的名稱,-root文件指定輸出的root文件的名稱,-chrom指定需要分析的染色體,默認情況下分析所有的染色體,這里我是測試,只分析了1號染色體。
這一步是按照固定大小的窗口對基因組進行劃分,統計每個窗口內的read depth, 代碼如下
cnvnator -root file.root -his 1000 -chrom 1 -d genome_fa_dir
-his參數指定窗口的大小,單位為bp, -d參數指定參考基因組fasta文件所在的目錄。
這一步是必須的,代碼如下
cnvnator -root file.root -stat 1000
-stat指定窗口的大小,和第二步的-his參數的取值相同。
這一步進行segmentation,代碼如下
cnvnator -root file.root -partition 1000
-partition指定窗口的大小,和第二步的-his參數的取值相同。
這一步分析每個segment區域的CNV, 代碼如下
cnvnator -root file.root -call 1000 > cnv.call.txt
-call指定窗口的大小,和第二步的-his參數的取值相同, 輸出結果默認打印到屏幕上,這里重定向到文件中。
輸出文件的內容如下
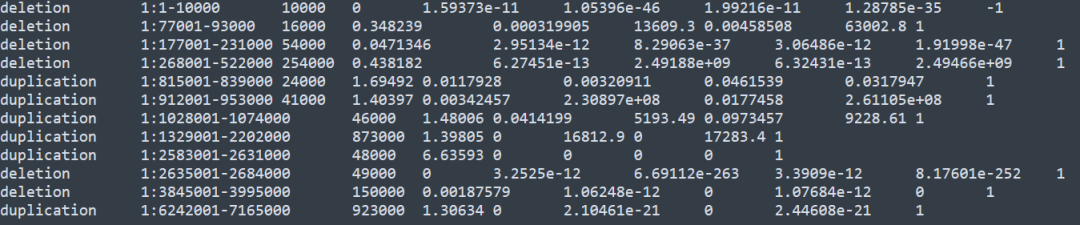
第一列為CNV的類型,包括了deletion和duplication兩種類型,第二列為CNV的染色體區域,第三列為CNV的長度,第四列為歸一化之后的read depth, 歸一化到0-1的范圍,第五列到第八列為不同的evalue值,最后一類為q0, 后五列對應的解釋如下

對于原始的cnv call的輸出,還可以通過軟件自帶的腳本轉換為VCF格式,代碼如下
cnvnator2VCF.pl cnv.call.txt >cnv.vcf
CNVnator的功能強大,運行速度快,更多用法請參考官方文檔。
到此,關于“怎么使用CNVnator進行CNV檢測”的學習就結束了,希望能夠解決大家的疑惑。理論與實踐的搭配能更好的幫助大家學習,快去試試吧!若想繼續學習更多相關知識,請繼續關注億速云網站,小編會繼續努力為大家帶來更多實用的文章!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。