您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
本篇文章給大家分享的是有關如何進行全基因組數據CNV分析,小編覺得挺實用的,因此分享給大家學習,希望大家閱讀完這篇文章后可以有所收獲,話不多說,跟著小編一起來看看吧。
除了利用aCGH和snp芯片來檢測CNV之外,也可以通過NGS數據來分析CNV, 比如全基因組和全外顯子測序。針對全基因組CNV的檢測,還針對開發了一種稱之為CNV_seq的測序策略,指的是低深度全基因組測序,只需要5X的測序深度,就可以有效的檢測CNV。
根據軟件的基本原理,可以分為以下4大類別,圖示如下
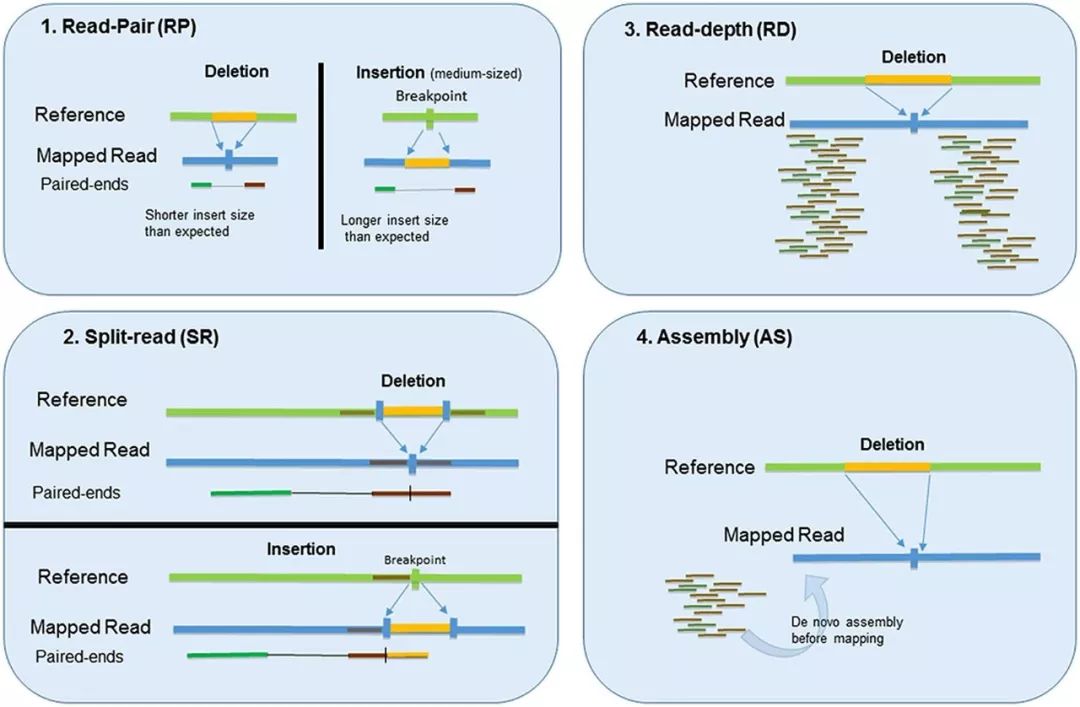
RP是最早出現的算法,利用雙端測序插入片段長度分布來檢測CNV, 也稱之為PEM,pair end mapping方法。雙端測序插入片段長度分布如下圖所示
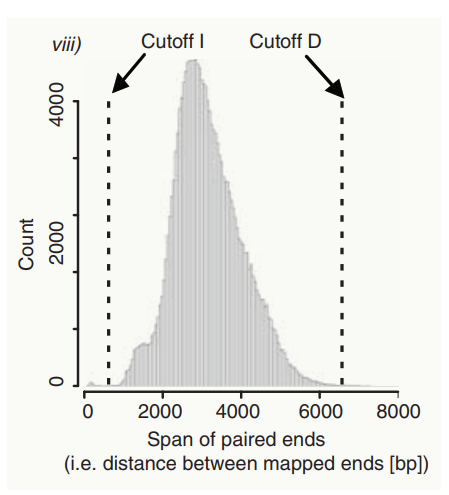
當插入片段長度過長或者過短時,都代表著基因組發生了結構變異,如上圖中的兩個閾值,圖示如下
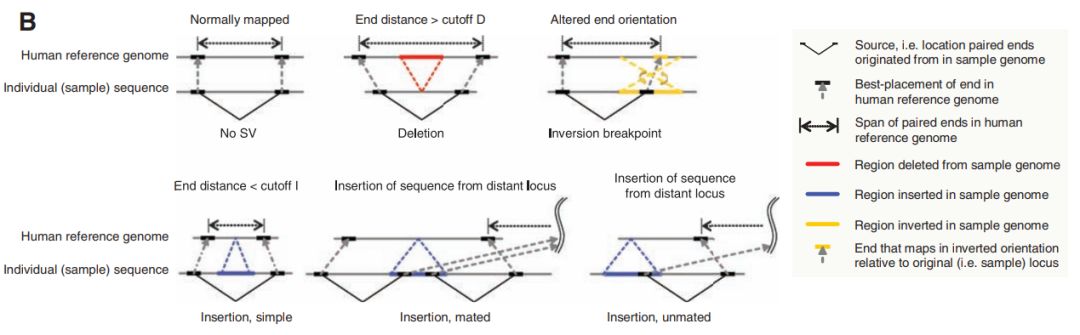
以上兩幅圖來自文獻Jan O. Korbel et al.Science 318, 420 (2007)
當計算出來的插入片段長度小于cutoff I時,說明相比reference, 實際檢測樣本中對應區域插入了部分堿基,相反地,如果計算出來的插入片段長度大于cutoff D時,說明相比reference, 實際檢測樣本對應區域插入了部分堿基。
受到測序讀長的影響,該方法適用于檢測中等長度的insertion和deletion, 對過小的插入不敏感,而且比較依賴比對的準確性,無法分析低復雜度的segmental duplication區域。
采用該策略的部分軟件列表如下
BreakDancer
PEMer
Ulysses
SR方法利用一端能夠比對,另外一端比對不上的reads來識別CNV。另外一端比對不上,可能是存在CNV, 通過將單獨的reads進行拆分,使其能夠正確比對到參考基因組上,拆分的點就是CNV的斷裂點。
只利用了單端reasd, 讀長進一步受到限制,所以該方法只適用于檢測小規模的插入和缺失,采用該策略的部分軟件列表如下
Pindel
PRISM
SVseq2
Gustaf
RD方法利用拷貝數和對應區域測序深度的相關性來進行分析,基本模型是缺失區域的測序深度相對低,而插入區域的測序深度相對高。該算法采用滑動窗口的方式,統計每個窗口內的測序深度分布,然后根據不同窗口測序深度的分布來預測CNV區域,圖示如下
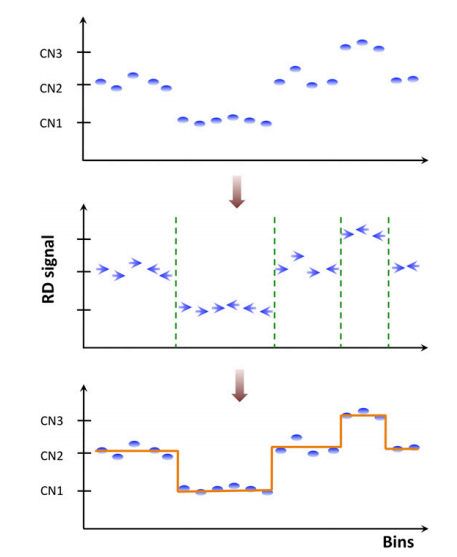
上圖來自文獻Genome Res. 2011. 21: 974-984
類似芯片中的log ratio值,在RD方法中,會根據區域對應的測序深度來判斷對應的CNV數目。在該類方法中,滑動窗口的大小對結果影響較大,當窗口很大時,一些長度很短的small cnv信號就會被掩蓋。
相比RP和SR兩種方法,RD可以進行CNV分型,明確CNV的數目,RP和SR只能檢測斷點的位置, 而且RD可以檢測大規模的CNV, 是目前較為主流的算法。采用該策略的部分軟件列表如下
CNVnator
ERDS
ReadDepth
CNVrd2
AS方法利用測序得到的短序列進行組裝,將組裝的contig與參考基因組進行比較,從而確定發生了結構變異的區域。組裝的精確依賴測序讀長和算法的準確度,而且組裝對硬件資源的消耗特別大,并不是一個理想的CNV檢測的算法,這里就不做過多的介紹了。
以上4種是最基本的算法理念,還有很多軟件會綜合其中的某幾種算法來檢測CNV, 比如speedseq中集成的lumpy軟件,綜合利用RP,SR, RD三種方式來檢測CNV。
比對準確性是基于NGS的策略檢測結果準確的前提,mapping的準確率和二代測序對基因組的覆蓋度都會影響到CNV的檢測結果,同時在計算測序深度時GC含量差異帶來的PCR擴增偏移,也需要進行校正,通過設置對照樣本,能夠有效的減少系統誤差的干擾,更好的進行CNV的檢測。
綜上所述,每種算法各有其優缺點,綜合使用多種策略有助于提高檢測結果的準確性和敏感性,同時設置對照樣本,可以更加有效的分析拷貝數的變化。
以上就是如何進行全基因組數據CNV分析,小編相信有部分知識點可能是我們日常工作會見到或用到的。希望你能通過這篇文章學到更多知識。更多詳情敬請關注億速云行業資訊頻道。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。