您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
今天就跟大家聊聊有關怎么使用R語言ggtree展示進化樹,可能很多人都不太了解,為了讓大家更加了解,小編給大家總結了以下內容,希望大家根據這篇文章可以有所收獲。
今天要模仿的圖片來自于論文 Core gut microbial communities are maintained by beneficial interactions and strain variability in fish。期刊是 Nature microbiology

今天重復的圖片是Figure1中的聚類樹圖
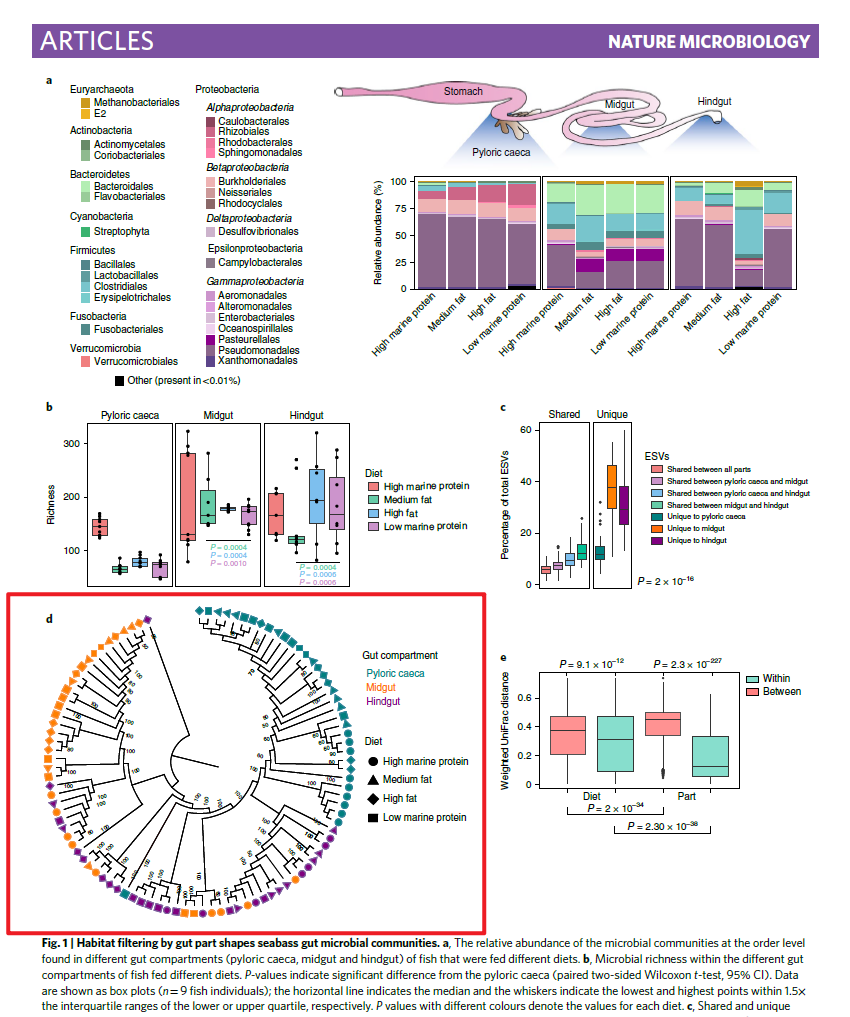
Hierarchical clustering dendrogram with jackknife support (numbers on the branches; only values above 50 are shown in the tree).
所以論文中實際的數據做的是聚類分析,而并不是進化樹。他這里做聚類分析也能夠獲得每個節點對應的支持率。這個如何實現我暫時還不知道。為了模仿這個圖,下面的輸入數據我直接使用進化樹文件了,因為構建進化樹的時候能夠很方便的獲得節點的支持率信息。
這里用到的數據集來自 網址 https://www.kuleuven.be/aidslab/phylogenybook/Data_sets.html
這本 The Phylogenetic Handbook second edition 不知道大家有沒有電子版可以分享呀!
mafft --auto ggtree_practice.fasta > ggtree_practice_aligned.fasta
iqtree -s ggtree_practice_aligned.fasta -bb 1000
得到樹文件ggtree_practice_aligned.fasta.treefile
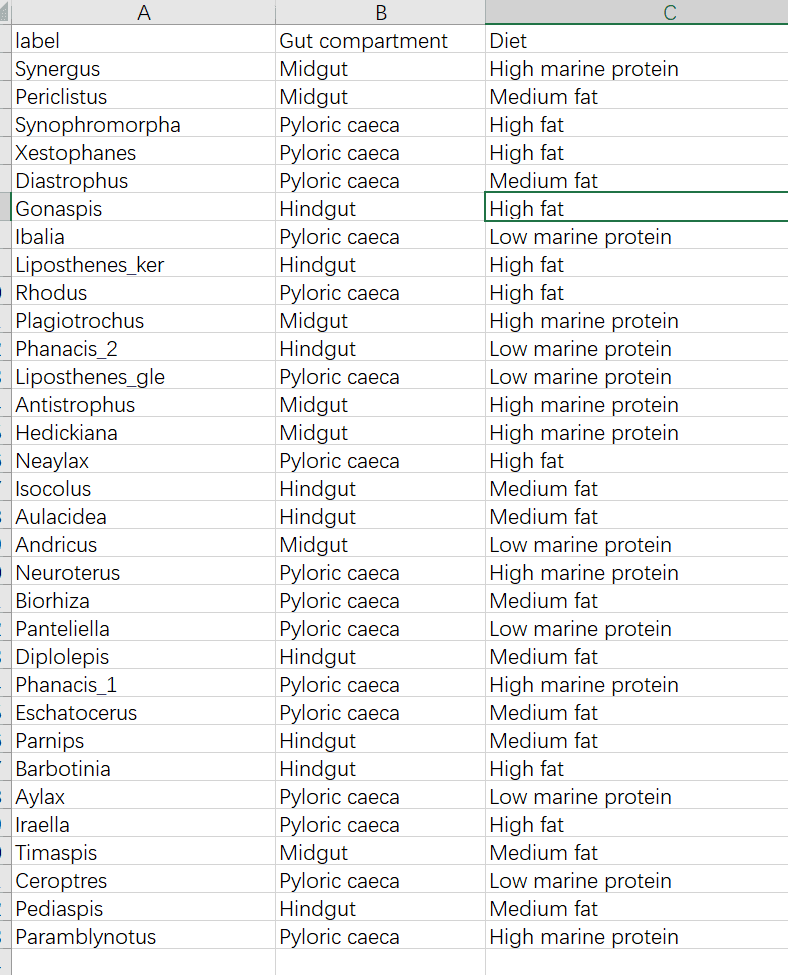
數據總共三列
第二列和第三列的列名就是圖例上想顯示什么就用什么
library(ggtree)
library(treeio)
library(tidytree)
treeio用來讀入進化樹tidytree用來將分組信息整合給進化樹ggtree用來可視化展示進化樹
tree<-read.newick("ggtree_practice_aligned.fasta.treefile",
node.label = "support")
d<-read.csv("ggtree_group_info.csv",header=T)
d
trs<-full_join(tree,d,by='label')
tree@data$support<-ifelse(tree@data$support<50,NA,tree@data$support)
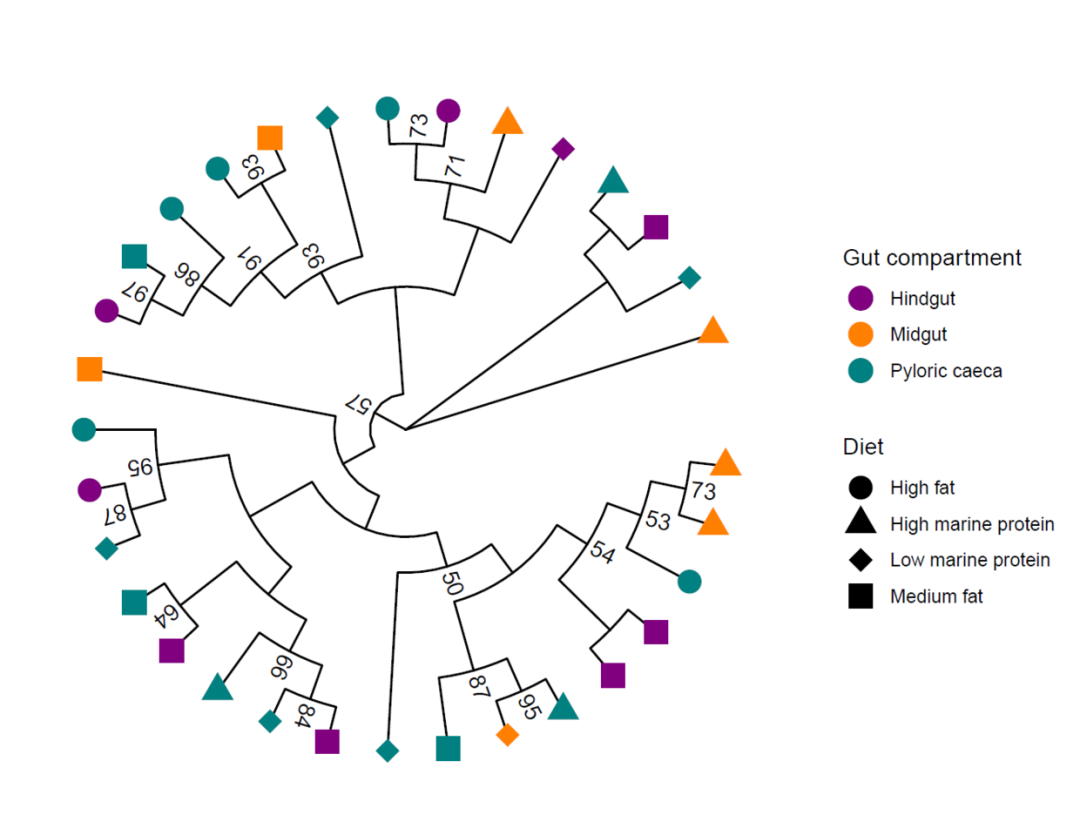
ggtree(trs,layout = "circular",branch.length = "none")+
#geom_tiplab(offset = 0.01)+
geom_tippoint(aes(shape=Diet,color=Gut.compartment),
size=5)+
scale_shape_manual(values = c(16,17,18,15))+
geom_text2(aes(label=support,angle=angle),hjust=-0.2)+
scale_color_manual(values = c("#800080","#ff8000","#008080"),
name="Gut_compartment")+
guides(color=guide_legend(order = 1))
 看完上述內容,你們對怎么使用R語言ggtree展示進化樹有進一步的了解嗎?如果還想了解更多知識或者相關內容,請關注億速云行業資訊頻道,感謝大家的支持。
看完上述內容,你們對怎么使用R語言ggtree展示進化樹有進一步的了解嗎?如果還想了解更多知識或者相關內容,請關注億速云行業資訊頻道,感謝大家的支持。免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。