您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
本篇內容主要講解“如何使用Beagle進行基因型填充”,感興趣的朋友不妨來看看。本文介紹的方法操作簡單快捷,實用性強。下面就讓小編來帶大家學習“如何使用Beagle進行基因型填充”吧!
軟件的官網如下
https://faculty.washington.edu/browning/beagle/beagle.html
相比同類軟件,該軟件有兩個顯著的優勢,第一個就是內存消耗小,第二個運行速度快。對于數據量如此大的reference panel數據,提出了一種新的數據存儲格式bref3。文章中比較了不同格式消耗的內存大小,結果如下
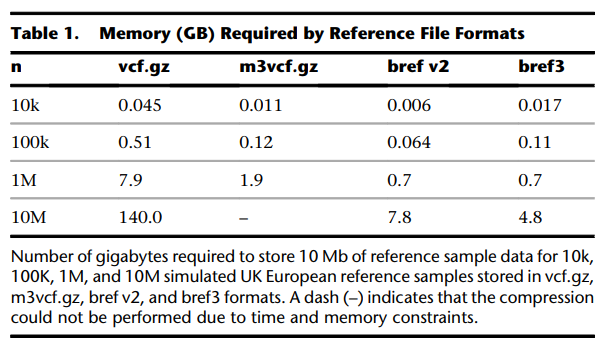
可以看到,bref3格式的內存消耗最小。關于運行時間的比較結果如下
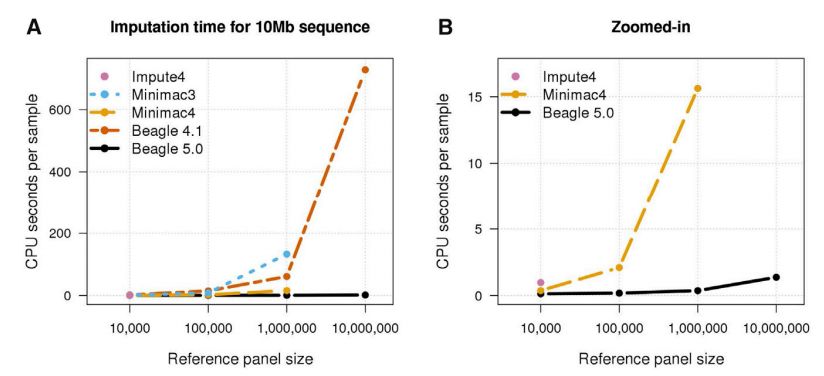
可以看到Beagle 5.0版本的運行時間最短。該軟件采用java語言進行開發,安裝簡單,直接下載jar文件即可。基本用法如下
java \
-jar beagle.24Aug19.3e8.jar \
ref=chr1.1kg.phase3.v5a.b37.bref3 \
map=plink.GRCh48.map \
gt=input.chr1.vcf.gz \
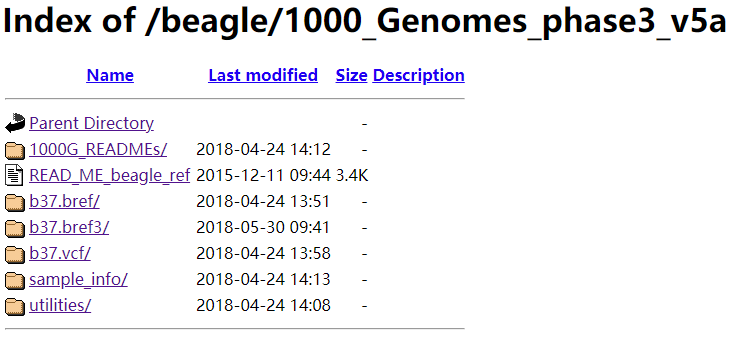
out=out.chr1.gtref參數指定reference panel的數據,支持vcf和bref3兩種格式,官網提供了1000G的數據供下載,鏈接如下
http://bochet.gcc.biostat.washington.edu/beagle/1000_Genomes_phase3_v5a/
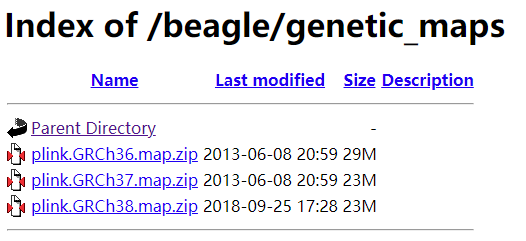
map參數指定參考基因組的連鎖圖譜,是一個可選參數,官網提供了human對應的連鎖圖譜,鏈接如下
http://bochet.gcc.biostat.washington.edu/beagle/genetic_maps/
到此,相信大家對“如何使用Beagle進行基因型填充”有了更深的了解,不妨來實際操作一番吧!這里是億速云網站,更多相關內容可以進入相關頻道進行查詢,關注我們,繼續學習!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。