您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章主要講解了“如何使用Minimac進行基因型填充”,文中的講解內容簡單清晰,易于學習與理解,下面請大家跟著小編的思路慢慢深入,一起來研究和學習“如何使用Minimac進行基因型填充”吧!
源代碼保存在github上,網址如下
https://github.com/statgen/Minimac4
為了減少內存消耗,和beagle軟件類似,minimac提出了一種名為M3VCF的格式,用來存儲referenc panel的單倍型信息,將單倍型劃分為不同的block, 示意如下
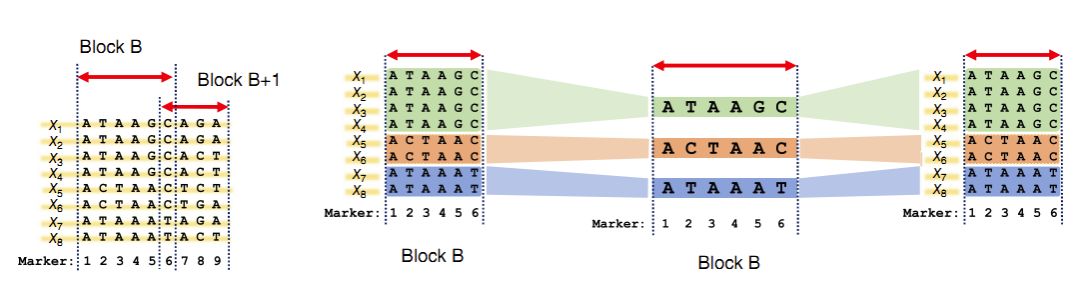
上圖表示的是9個SNP位點構成的8種單倍型,minimac會根據染色體位置劃分成不同的block區間,識別block區間內的unique haplotypes。兩個鄰近的block區域必須有一個重疊的位點,圖中的9個位點劃分成了兩個block, 1-6號位點對應block B, 6到9號位點對應block B+1。在block B中,有3種唯一的單倍型,對應三種不同顏色。
M3VCF的內容示意如下
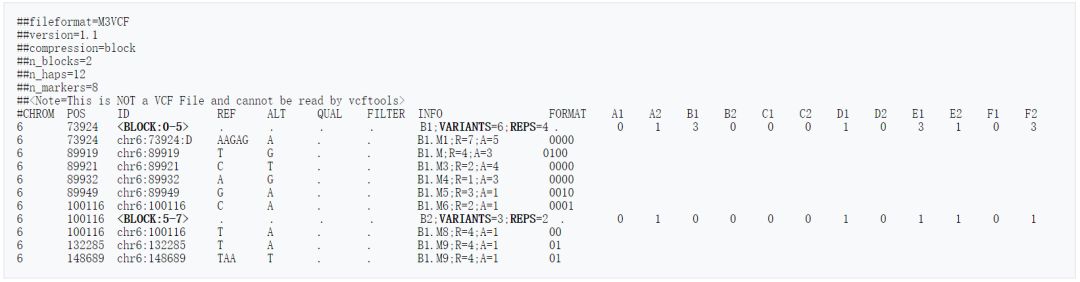
和VCF格式類似,在每個blcok的開頭會有一行用于顯示block的信息,包含的變異位點數VARIANTS,unique haplotype的個數REPS等,詳細的介紹請參考以下鏈接
https://genome.sph.umich.edu/wiki/M3VCF_Files
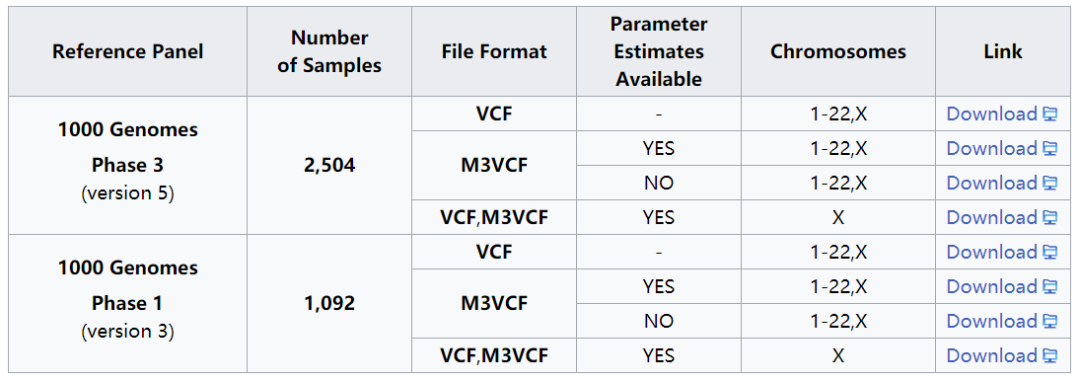
相比VCF格式,該格式磁盤占用小,讀取速度更快,內存消耗小。官網提供了1000G的reference panel供下載,鏈接如下
https://genome.sph.umich.edu/wiki/Minimac4#Reference_Panels_for_Download
該軟件推薦的基因型填充pipeline步驟如下
study樣本分型結果的質量控制,參考GWAS的質控條件
對質控后的分型結果進行pre-phasing, 支持MACH和shapeit兩個軟件的結果
將pre-phasing的結果轉化成VCF格式
選擇reference panel,推薦1000G或者HRC
進行填充
實際操作中典型的用法如下
以MACH為例,用法如下
mach2 \
-d Gwas.chr20.Unphased.dat \
-p Gwas.chr20.Unphased.ped \
--rounds 20 \
--states 200 \
--phase \
--interim 5 \
--sample 5 \
--prefix Gwas.Chr20.Phased.OutputMACH要求輸入的分型結果格式為dat和ped格式,詳細的解釋參見以下鏈接
http://csg.sph.umich.edu/abecasis/Merlin/tour/input_files.html
通過MACH2VCF進行格式轉換,用法如下
mach3VCF \
--haps Gwas.Chr20.Phased.Output.hap \
--snps Gwas.Chr20.Phased.Output.snps \
--prefix Gwas.Chr20.Phased.Output.VCF.Format該腳本可以從以下鏈接下載
ftp://share.sph.umich.edu/minimac3/Mach3VCF.v1.Source.Binary.tar.gz
采用minimac4進行填充,用法如下
minimac4 \
--refHaps refPanel.m3vcf \
--haps Gwas.Chr20.Phased.Output.VCF.Format.vcf \
--prefix testRun \
--cpus 5minimac4會自動將染色體拆分成不同的區間,在結合多線程,可以大大提高運行速度。
感謝各位的閱讀,以上就是“如何使用Minimac進行基因型填充”的內容了,經過本文的學習后,相信大家對如何使用Minimac進行基因型填充這一問題有了更深刻的體會,具體使用情況還需要大家實踐驗證。這里是億速云,小編將為大家推送更多相關知識點的文章,歡迎關注!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。