您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
本篇內容主要講解“如何使用homer進行peak注釋”,感興趣的朋友不妨來看看。本文介紹的方法操作簡單快捷,實用性強。下面就讓小編來帶大家學習“如何使用homer進行peak注釋”吧!
homer軟件集成了許多的功能,包括peak calling, peak注釋,motif分析等等,通過這一個軟件,就可以完成chip_seq的絕大部分分析內容,不可謂不強大。本文主要介紹這個軟件進行peak注釋的用法。
在homer中通過annotatePeaks.pl這個腳本進行peak的注釋,分為以下兩步
homer內置了許多物種的注釋信息供我們下載,通過以下命令可以查看所有內置的物種
perl configureHomer.pl --list
其中GENOMES部分對應的就是內置支持的物種,部分內容展示如下
GENOMES
v5.10 hg19 v6.0 human genome and annotation for UCSC hg19
+ mm10 v6.0 mouse genome and annotation for UCSC mm10
- sacCer3 v6.0 yeast genome and annotation for UCSC sacCer3
- panTro5 v6.0 human genome and annotation for UCSC panTro5以hg19為例,下載方式如下
perl configureHomer.pl -install hg19
下載的信息保存在homer安裝目錄的data目錄下,以hg19為例,在data/genome/hg19目錄下,文件列表如下
├── chr1.fa
├── chr2.fa
├── chr3.fa
├── ...fa
├── chrom.sizes
├── conservation
├── hg19.annotation
├── hg19.aug
├── hg19.basic.annotation
├── hg19.full.annotation
├── hg19.miRNA
├── hg19.repeats
├── hg19.rna
├── hg19.splice3p
├── hg19.splice5p
├── hg19.stop
├── hg19.tss
├── hg19.tts
└── preparsed包含了參考基因組的fasta序列以及不同區域的區間文件。
hg19.basic.annotation內容如下
Intergenic chr1 1 10873 + N 1900000000
promoter-TSS (NR_046018) chr1 10874 11974 + P 1
non-coding (NR_046018, exon 1 of 3) chr1 11975 12227 + pseudo 125025
intron (NR_046018, intron 1 of 2) chr1 12228 12612 + I 810684
non-coding (NR_046018, exon 2 of 3) chr1 12613 12721 + pseudo 125026
intron (NR_046018, intron 2 of 2) chr1 12722 13220 + I 810684
non-coding (NR_046018, exon 3 of 3) chr1 13221 13361 + pseudo 125027同時在data/accession目錄下,還有參考基因組對應的基因注釋文件。
human2gene.tsv記錄了基因的ubigene id, gene symbol等信息,內容如下所示
ADE73044 3107 Hs.656020 NM_002117 ENSG00000204525 HLA-C
ENSG00000113163 10087 Hs.270437 NM_005713 ENSG00000113163 COL4A3BP
DB065460 9947 Hs.132194 NM_005462 ENSG00000155495 MAGEC1
ENSP00000282466 285313 Hs.58561 NM_178822 ENSG00000152580 IGSF10
DB029361 22849 Hs.131683 NM_014912 ENSG00000107864 CPEB3
XP_016877211 87 Hs.235750 NM_001102 ENSG00000072110 ACTN1
EAW77897 56965 Hs.270244 NM_020213 ENSG00000137817 PARP6human.description記錄表了基因的功能描述,類別等信息,示意如下
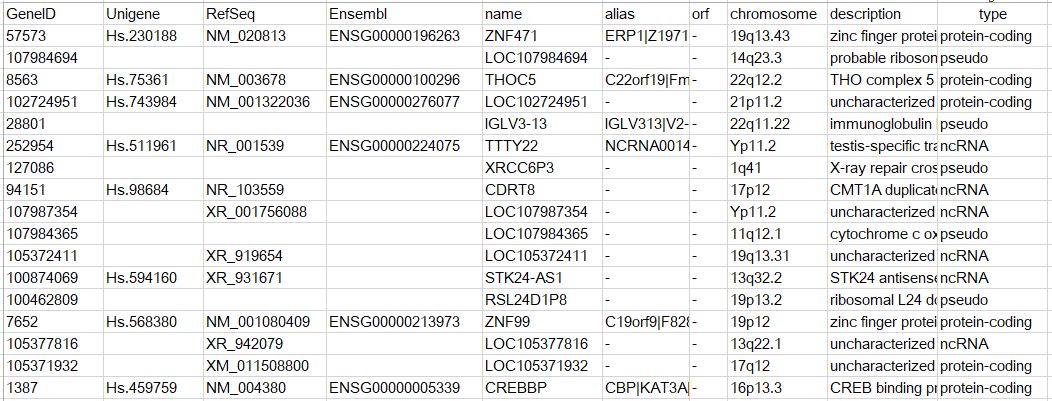
用法如下
annotatePeaks.pl peak.bed hg19 > peak.annotation.xls
第一個參數為peak的bed文件,第二個參數為參考基因組的名稱。輸出結果如下所示
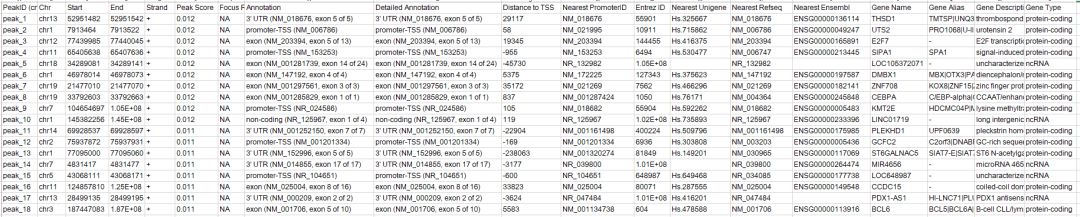
注釋的內容包含兩個部分,第一部分是距離peak區間最近的轉錄起始位點TSS,第二部分是對peak在基因組區域的分布,比如TSS,TTS,3’UTR,5’UTR等區域。
到此,相信大家對“如何使用homer進行peak注釋”有了更深的了解,不妨來實際操作一番吧!這里是億速云網站,更多相關內容可以進入相關頻道進行查詢,關注我們,繼續學習!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。