您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這期內容當中小編將會給大家帶來有關環狀RNA注釋工具circAnno怎么理解,文章內容豐富且以專業的角度為大家分析和敘述,閱讀完這篇文章希望大家可以有所收獲。
在利用RNA_seq數據預測環狀RNA時,大多數情況下只能夠得到環狀RNA的基因組位置,包括頭尾的染色體位置,正負鏈等信息,而沒有環狀RNA對應的來源基因,序列等信息,這些信息都需要我們通過和已有的線性RNA的數據比對得到,這一步在分析中稱之為環狀RNA的注釋。
對于環狀RNA注釋,不同團隊有不同的做法,核心就是利用環狀RNA的基因組位置和已知的轉錄本去比較,確定是否和已知轉錄本有重疊,從而確定來源基因和對應的轉錄本。
在starBase數據庫中,提供了一系列環狀RNA注釋工具
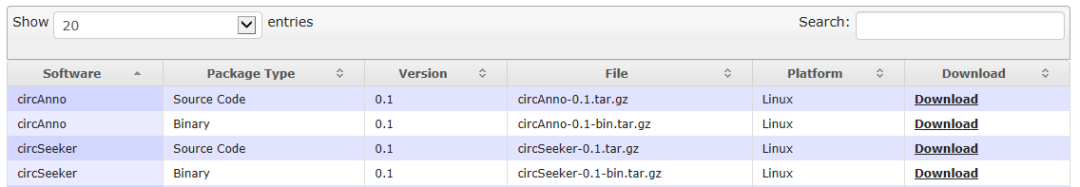
其中circSeeker和circAnno就是用來進行環狀RNA注釋的工具
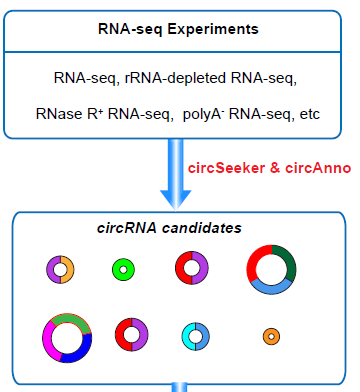
circAnno需要兩個輸入文件,一個為環狀RNA對應的bed格式的文件,另外一個為線性RNA對應的bed12格式的文件,以circBase數據庫中的環狀RNA為例來看下這個軟件的具體用法。
wget http://www.circbase.org/download/hsa_hg19_circRNA.bed cut -f1-6 hsa_hg19_circRNA.bed > circRNA.bed6
利用table browser進行下載,圖示如下
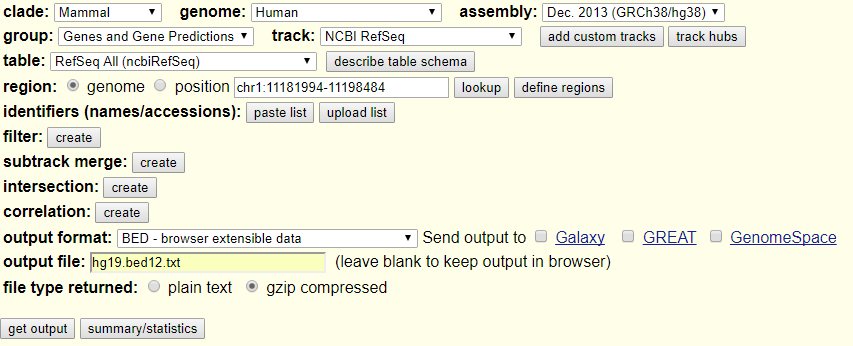
circAnno hg19.bed12.txt circRNA.bed6 > annotation.xls
輸出結果內容如下
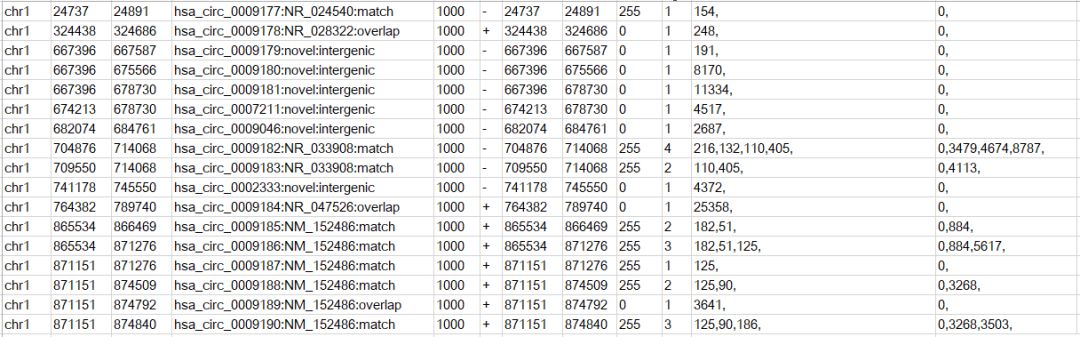
前六列的內容就是環狀RNA的bed文件內容,只不過在第四列的基礎上增加了轉錄本注釋信息,match表示和轉錄本的exon完全匹配,overlap表示重疊,intergenic代表是基因間區。最后三列分別代表重疊區域的外顯子個數,外顯子長度和外顯子的起始位置,和bed12文件中的內容一致。
上述就是小編為大家分享的環狀RNA注釋工具circAnno怎么理解了,如果剛好有類似的疑惑,不妨參照上述分析進行理解。如果想知道更多相關知識,歡迎關注億速云行業資訊頻道。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。