您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章主要介紹R語言如何實現可視化展示gff3格式基因組注釋文件,文中介紹的非常詳細,具有一定的參考價值,感興趣的小伙伴們一定要看完!
使用R語言的 ggbio 這個包可視化gff3格式的基因組注釋文件
我用到的文件是NCBI下載的擬南芥注釋文件,為了減小計算壓力,我只用到了gff文件的前119行,兩個基因。
用到的函數是 GenomicFeatures R包中的 **makeTxDbFromGFF()**函數
library(GenomicFeatures)
txdb<-makeTxDbFromGFF(file="practice.gff",format="gff3")
用到的 ggbio 這個包中的 **autoplot()**這個函數
library(ggbio)
autoplot(txdb,
which=GRanges("CP002684.1", IRanges(100, 9000)),
names.expr = "gene_id")+
theme_bw()
結果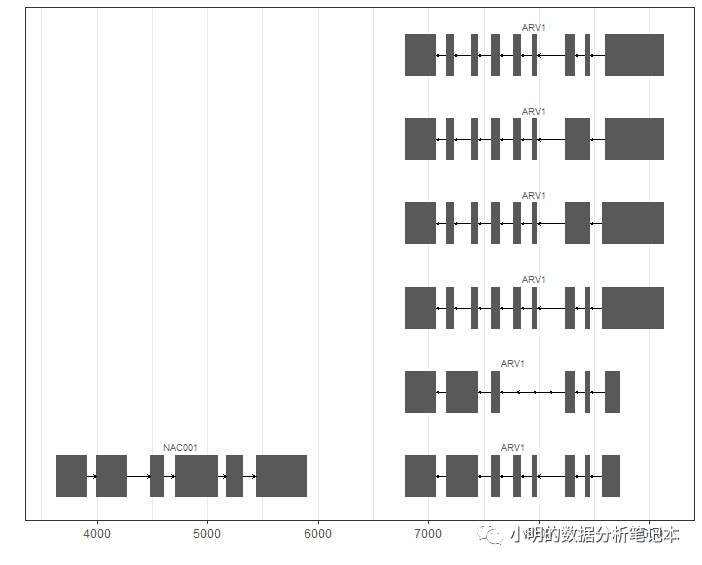
可以通過fill參數設置不同的顏色
autoplot(txdb,
which=GRanges("CP002684.1", IRanges(100, 9000)),
names.expr = "gene_id",fill="red")+
theme_bw()
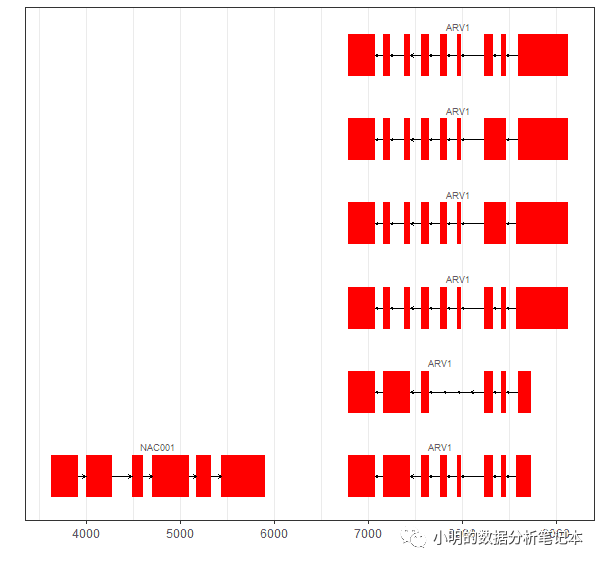
不同的基因填充不同的顏色
autoplot(txdb,
which=GRanges("CP002684.1", IRanges(100, 9000)),
names.expr = "gene_id",aes(fill=gene_id))+
theme_bw()
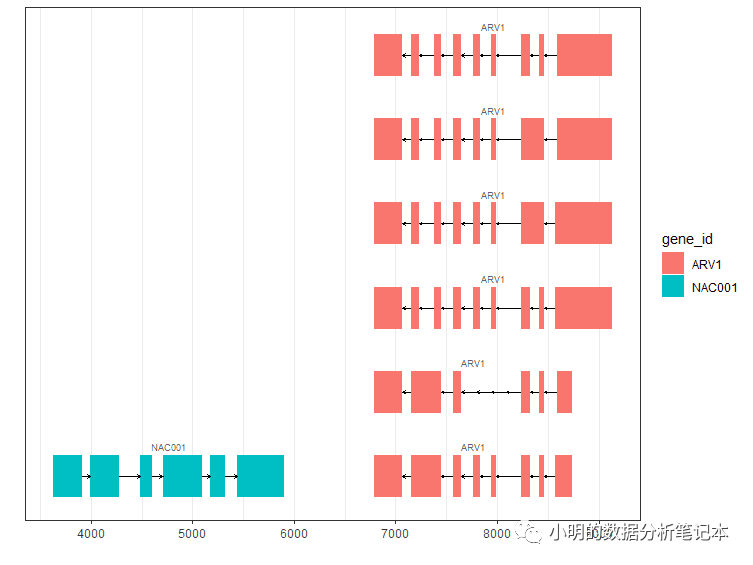
現在還不知道如何給同一個基因不同的部分(utr,exon,intron)等填充不同的顏色 還有就是 makeTxDbFromGFF() 函數讀入的數據存儲格式還沒搞懂
開頭提到的參考資料里有一幅圖將 reads數量, 覆蓋度的折線圖,vcf文件的結果,gff可視化的結果畫到了一起,做基因組重測序分析應該會用得到。這里暫時不重復了。等用到的時候再說。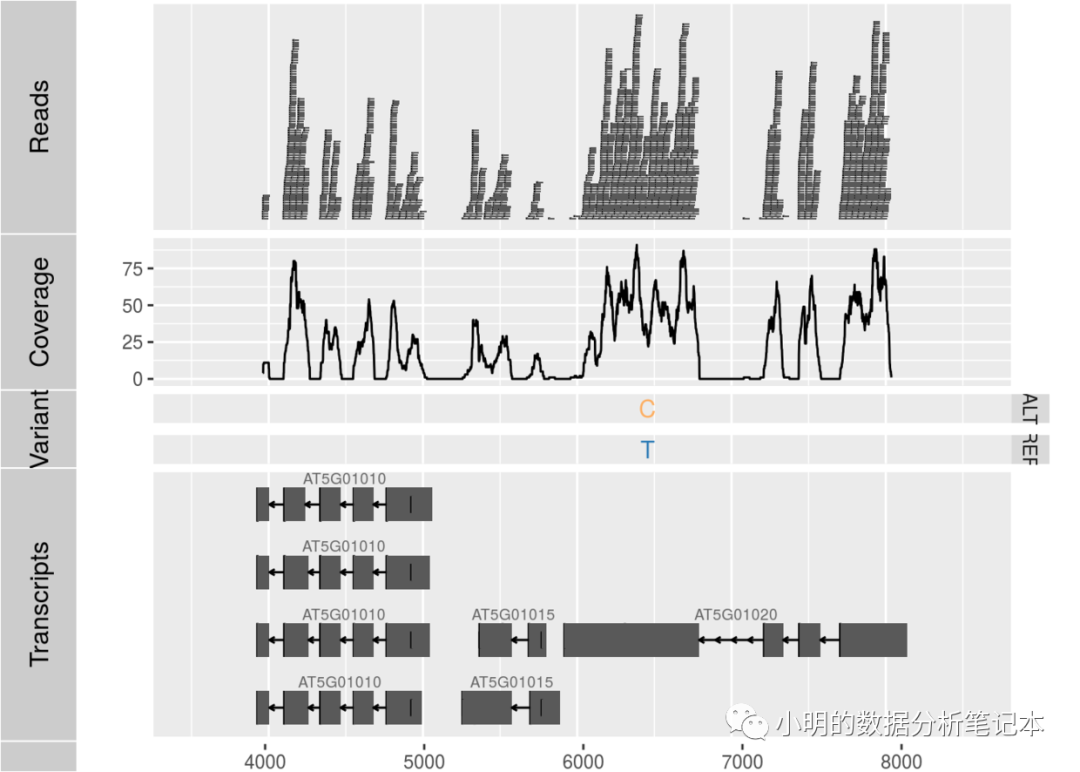
以上是“R語言如何實現可視化展示gff3格式基因組注釋文件”這篇文章的所有內容,感謝各位的閱讀!希望分享的內容對大家有幫助,更多相關知識,歡迎關注億速云行業資訊頻道!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。