您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章將為大家詳細講解有關HiCPlotter工具有什么用,小編覺得挺實用的,因此分享給大家做個參考,希望大家閱讀完這篇文章后可以有所收獲。
HiCPlotter是一款命令行工具,用來展示Hi-C的交互矩陣。除了基本的用熱圖展示交互矩陣外,還支持添加基因結構,chip_seq等二維數據的注釋信息,網址如下
https://github.com/kcakdemir/HiCPlotter
對于交互矩陣,支持多種格式
Hi-C交互矩陣以bin為單位,每行和每列都代表一個bin, 對應單元格的數值反映兩個bin之間相互作用的頻率。通用格式如下所示
Bin1 Bin2 Bin3 Bin4 Bin5 Bin6
7.85957 4.80329 11.4766 9.57416 4.5288 8.55022
8.61621 4.98956 2.35654 5.69483 11.1187 10.1322
4.06803 4.07801 7.98047 2.59144 6.3851 7.74306
4.52869 2.70624 8.94544 4.29185 8.29491 8.38257#開頭的行以及第一行都被當做是注釋信息,會被忽略掉。
HiC-Pro為了解決傳統的交互矩陣太大的問題,專門制定了一種新的格式,如下所示
1050 1586 1
1050 1589 1
1050 1590 1 (jumps to 1612)
1050 1612 2共三列,前兩列對應bin的編號,第三列的數字代表兩個bin之間交互作用的頻率,不存在相互作用的bin就不會記錄在該文件之中。除了這個文件,還需要下列文件
chr1 20960000 20980000 1049
chr1 20980000 21000000 1050
chr1 21000000 21020000 1051
chr1 21020000 21040000 1052
chr1 21040000 21060000 1053
chr1 21060000 21080000 1054
chr1 21080000 21100000 1055給出了每個bin編號對應的染色質位置,通過這兩個文件就可以完整描述描述染色質交互信息了。
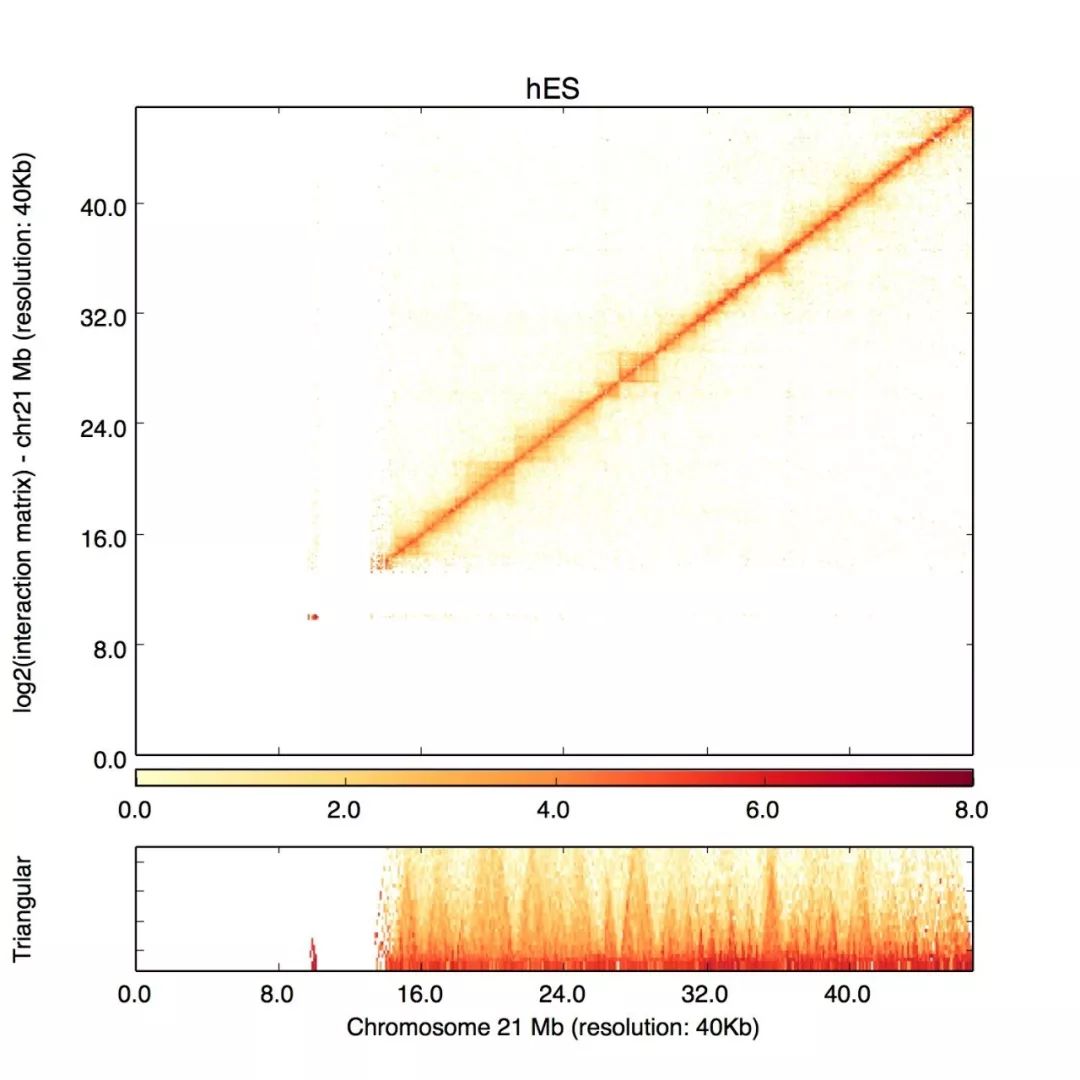
軟件的基本用法如下
python 、\
HiCPlotter.py \
-f matrix.txt \
-n hES \
-chr chr21 \
-r 40000 \
-o default1 \-f參數指定輸入的Hi-C交互矩陣,-n指定交互矩陣熱圖的標題名稱,-chr指定要畫的染色質的名稱,-r指定對應的分辨率,-o指定輸出文件的前綴, 結果示意如下
對于HiC-Pro的輸出格式,用法如下
python \
HiCPlotter.py \
-f matrix.txt \
-tri 1 \
-bed bin.bed \
-chr chr7 \
-o Example \
-r 40000 \
-n hES-tri 1聲明輸入的交互矩陣的格式是HiC-Pro的輸出格式,-bed指定bin編號對應的染色質位置文件,其他參數和基本用法相同。
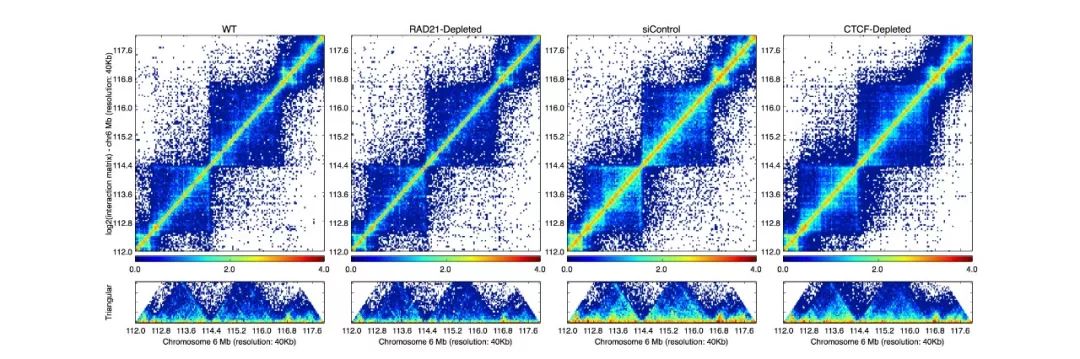
該軟件支持同時可視化多個交互矩陣,用法如下
python \
HiCPlotter.py \
-f matrix1.txt matrix2.txt matrix3.txt \
-n name1 name2 name3 \
-chr chr6 \
-r 40000 \
-o example對應的-f和-n參數為空格分隔的多個參數,結果示意如下
除了上述基本用法,還可以添加注釋信息,比如基因結構,對應chip_seq數據等,結果示意如下
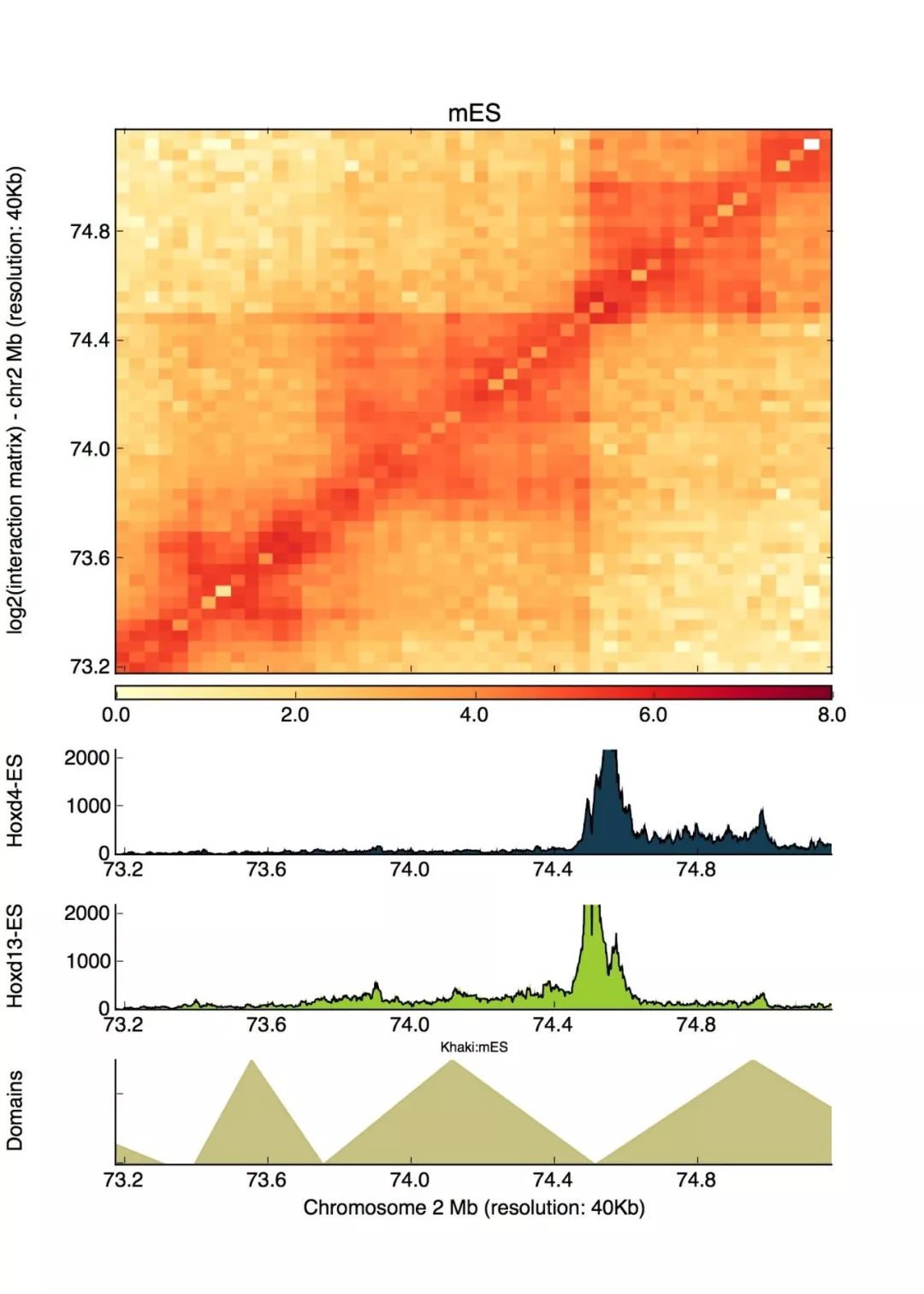
此時需要輸入各種注釋信息對應的文件,對于不同的注釋信息,要求的格式不同,更多的細節請參考官方文檔。
關于“HiCPlotter工具有什么用”這篇文章就分享到這里了,希望以上內容可以對大家有一定的幫助,使各位可以學到更多知識,如果覺得文章不錯,請把它分享出去讓更多的人看到。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。