您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
小編給大家分享一下Haplotype Reference Consortium數據庫有什么用,相信大部分人都還不怎么了解,因此分享這篇文章給大家參考一下,希望大家閱讀完這篇文章后大有收獲,下面讓我們一起去了解一下吧!
在進行基因型填充時,reference panel的選擇對填充結果的影響非常大,HapMap包含了3百多萬個SNP位點,420個單倍型,1000G包含了8千多萬個位點,5008個單倍型。除了這兩個常用的reference panel外,還有很多大型的人類基因組測序項目,比如UK10K等等。reference panel包含的單倍型越多,填充的準確率越高,涵蓋的SNP位點越多,填充后可以用于GWAS分析的位點就越多,可以更加有效的挖掘關聯信號。
Haplotype Reference Consortium簡稱HRC, 整合了來自UK10K, 1000G等多個項目的結果,構建了一個包含3千多萬個SNP位點,64976個單倍型的reference panel, 對應的文章發表在nature genetics上,鏈接如下
https://www.nature.com/articles/ng.3643

在文章中比較了不同reference panel對填充準確率的影響,結果如下
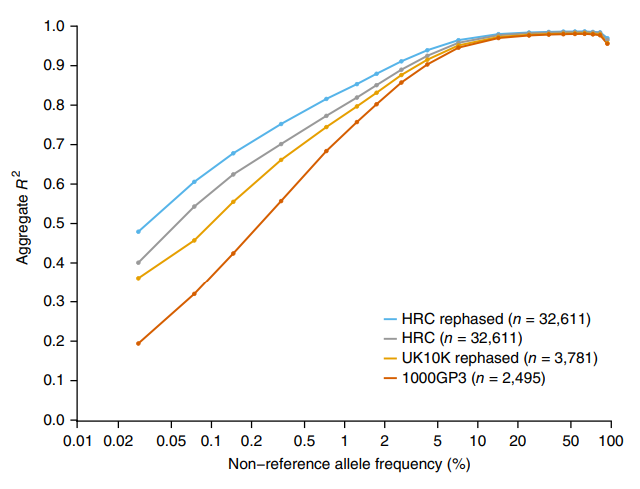
橫坐標為study樣本中不在reference panel中的SNP位點比例, 縱坐標為填充的準確率,可以看到HRC的填充準確率明顯高于UK10K和1000G。另外,對于reference panel對于GWAS結果的影響也進行了比較,結果如下
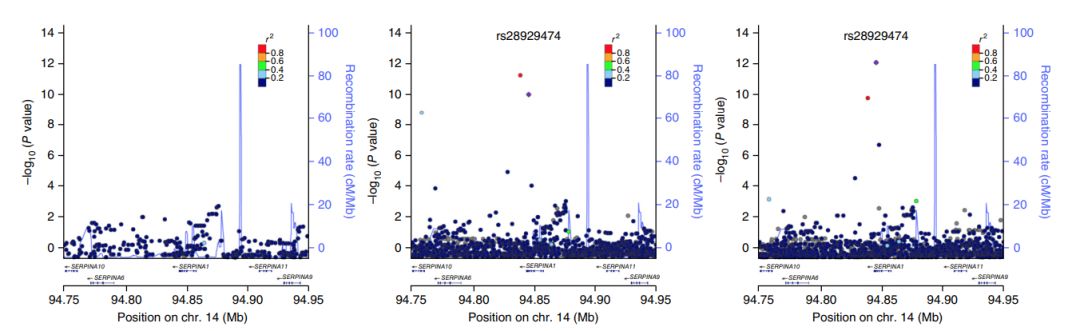
展示的是14號染色體上的曼哈頓圖,rs28929474是陽性結果,從左側到右側,分別對應hapmap, 1000G和HRC。使用HapMap填充,沒有識別到該陽性位點,1000G和HRC都識別到了該陽性位點。
HRC官網鏈接如下
http://www.haplotype-reference-consortium.org/
包含的基因組測序項目如下
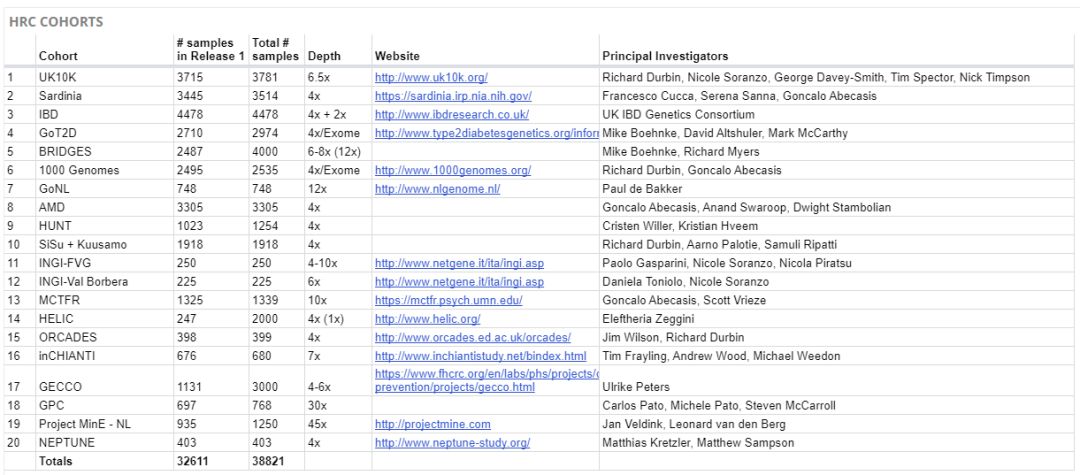
大部分為低深度全基因組測序的結果,共包含了32611個樣本,遺憾的是,該數據庫的信息并沒有完全公開,目前只有通過兩個在線網站,可以使用該數據庫進行基因型填充,網址如下
https://imputation.sanger.ac.uk/
https://imputationserver.sph.umich.edu/
在EBI中,包含了該數據庫的一個子集供下載,網址如下
https://www.ebi.ac.uk/ega/studies/EGAS00001001710
對于基因型填充而言,構建更大規模單倍型數據庫是提高準確率的有效方法,采用HRC數據庫,可以有效提供填充準確率。
以上是“Haplotype Reference Consortium數據庫有什么用”這篇文章的所有內容,感謝各位的閱讀!相信大家都有了一定的了解,希望分享的內容對大家有所幫助,如果還想學習更多知識,歡迎關注億速云行業資訊頻道!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。