您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章主要講解了“R語言層次聚類與聚類樹怎么使用”,文中的講解內容簡單清晰,易于學習與理解,下面請大家跟著小編的思路慢慢深入,一起來研究和學習“R語言層次聚類與聚類樹怎么使用”吧!
在生態學研究當中,有些環境中的對象是連續(或者離散)的,而有些對象是不連續的,聚類的目的是識別在環境中不連續的對象子集,從而探索隱藏在數據背后的屬性特征。聚類分析主要處理那些對象有足夠的相似性被歸于一組,并且確定組與組之間的差異或分離程度。聚類可以分為特征聚類(Vector Clustering)和圖聚類(Graph Clustering)。特征聚類是指根據對象的特征向量矩陣來計算距離或者相關性來實現聚類,例如各種層次聚類和非層次聚類。而圖聚類則針對的是復雜網絡數據,有隨機游走、貪心策略、標簽傳播等算法等。
根據對象歸屬方法,有以下兩種:
⑴硬劃分,也即將總體劃分為不同的部分,每個對象或者變量只能歸屬于某一組(身份信息為0或1)。
⑵模糊劃分,對象歸屬身份信息可以是連續的,也即身份信息可以是0到1中間的任意值。
聚類的結果可以輸出為無層級分組,也可以是具有嵌套結構的層次聚類樹。非約束的聚類分析只是一種數據劃分,不是典型的統計方法,因此不必進行統計檢驗,但是約束的聚類分析(多元回歸樹)需要進行統計檢驗。
⑴單連接聚合聚類
單連接聚合聚類(singlelinkage agglomerative clustering)也稱作最近鄰分類(nearestneighbour sorting),依據最短的成對距離或最大相似性來依次連接對象直到連接完畢,兩個組之間最近的兩個對象之間距離即為組的距離。此方法一般使用弦距離矩陣(歐氏距離)進行分析,在hclust()函數中為"single"。
⑵完全連接聚合聚類
完全連接聚合聚類(completelinkage agglomerative clustering)也稱作最遠鄰分類(furthestneighbour sorting),依據最遠距離決定一個對象或者一個組是否與另一個組聚合,所有對象之間的距離必須完全計算然后進行比較。単連接聚合聚類更容易體現數據的梯度,而完全連接聚合聚類分類組之間差異更加明顯。在在hclust()函數中為"complete"。
⑶平均聚合聚類
平均聚合聚類(averageagglomerative clustering)是一類基于對象之間平均相異性或者聚類簇形心(centroid)的進行聚類的方法。一個對象加入一個組依據的是這個對象與這個組成員的平均距離。在hclust()函數中有等權重算術平均聚類"average"(UPGMA)、不等權重算術平均聚類"mcquitty"(WPGMA)、等權重形心聚類"centroid"(UPGMC)、不等權重形心聚類"median"(WPGMC)四種方法。在生態學中Bray-Curtis距離矩陣一般使用方法"average"進行分析,其聚類樹結構介于單連接和完全連接聚類之間。
⑷最小方差聚類
Ward最小方差聚類是一種基于最小二乘法線性模型準則的聚類方法。分組的依據是使組內距離平方和(方差)最小化,由于使用了距離的平方,常常使聚類樹基部過于膨脹,可取平方根再進行可視化。在hclust()函數中有"ward.D"、"ward.D2"兩種方法。
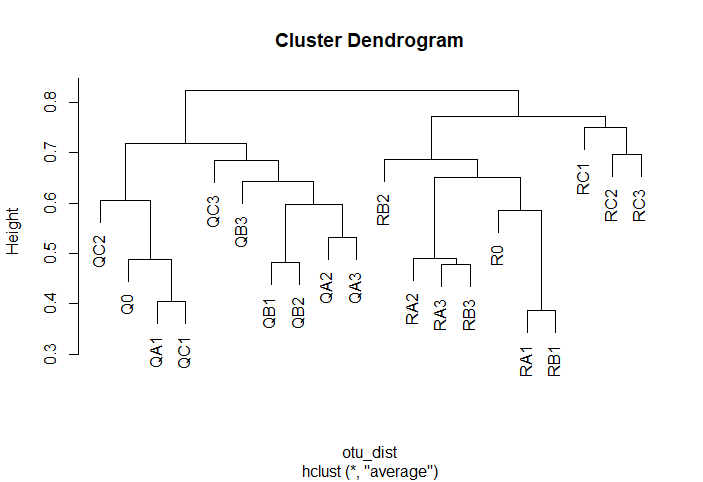
#讀取群落數據并計算Bray-Curtis距離矩陣data=read.table(file="sample.subsample.otu_table.txt", header=T, check.names=FALSE)rownames(data)=data[,1]data=data[,-1]library(vegan)data=decostand(data, MARGIN=2, "total")otu=t(data)otu_dist=vegdist(otu, method="bray", diag=TRUE, upper=TRUE, p=2)#進行聚類分析并作圖hclust=hclust(otu_dist, method="average")plot(hclust)
聚類樹如下圖所示:
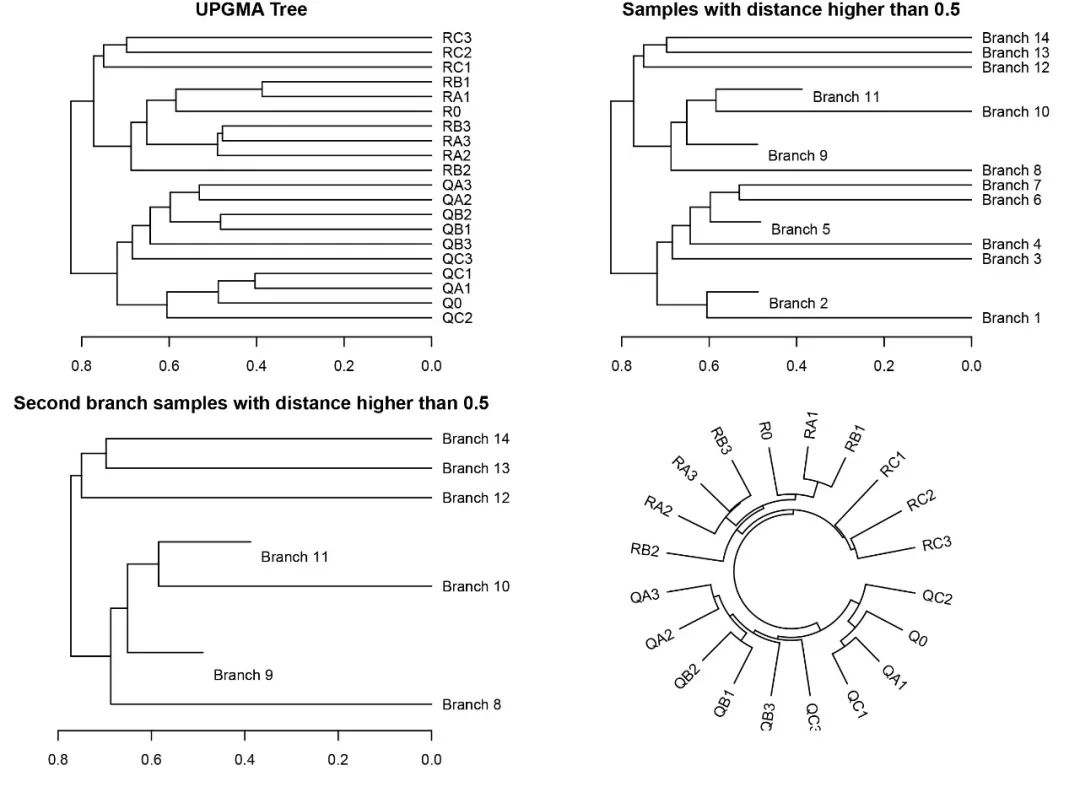
library(dendextend)library(circlize)tree=as.dendrogram(hclust)par(mfrow=c(2,2), mar=c(3,3,1,5), cex=0.7)plot(tree, horiz=TRUE, main="UPGMA Tree")#隱藏平均距離小于0.5的對象(類群)plot(cut(tree, h=0.5)$upper, horiz=TRUE, main="Samples with distance higher than 0.5")#在上一步基礎上篩選第二個分類簇plot(cut(tree, h=0.5)$upper[[2]], horiz=TRUE, main="Second branch samples with distance higher than 0.5")circlize_dendrogram(tree)
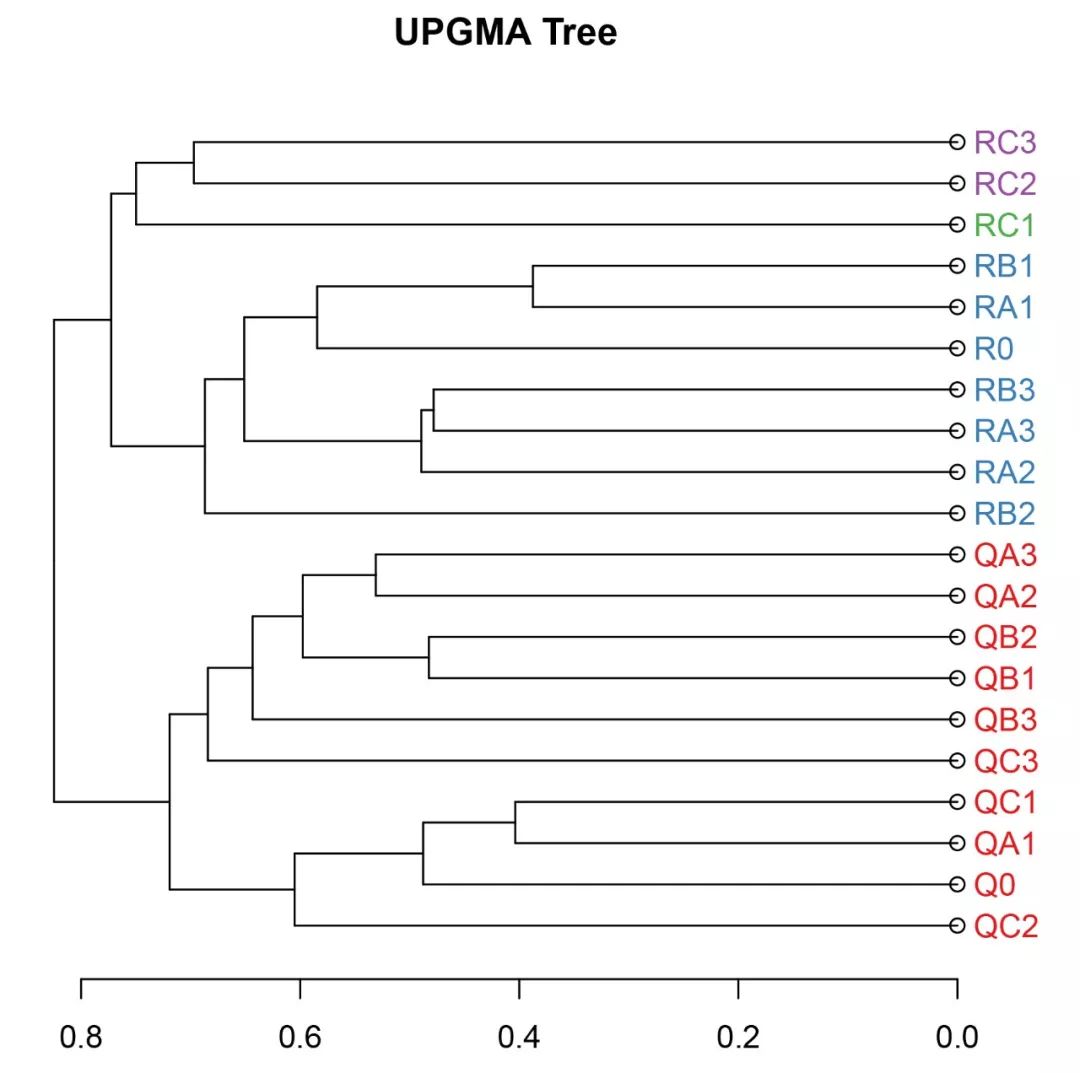
par(mfrow=c(1,1), mar=c(3,3,3,5), cex=1)#讀取群落數據并計算Bray-Curtis距離矩陣data=read.table(file="sample.subsample.otu_table.txt", header=T, check.names=FALSE)rownames(data)=data[,1]data=data[,-1]library(vegan)data=decostand(data, MARGIN=2, "total")otu=t(data)otu_dist=vegdist(otu, method="bray", diag=TRUE, upper=TRUE, p=2)#進行聚類分析并作圖hclust=hclust(otu_dist, method="average")library(dendextend)library(RColorBrewer)hcd=as.dendrogram(hclust)labelColors=brewer.pal(n=4, name="Set1")#聚類分組,預設聚類簇數目為4clusMember=cutree(hcd, 4)#自定義函數,根據聚類結果進行著色colLab=function(n) { if (is.leaf(n)) { a=attributes(n) labCol=labelColors[clusMember[which(names(clusMember)==a$label)]] attr(n, "nodePar")=c(a$nodePar, lab.col=labCol) } n}clusDendro=dendrapply(hcd, colLab)plot(clusDendro, main ="UPGMA Tree", type="rectangle", horiz=TRUE)
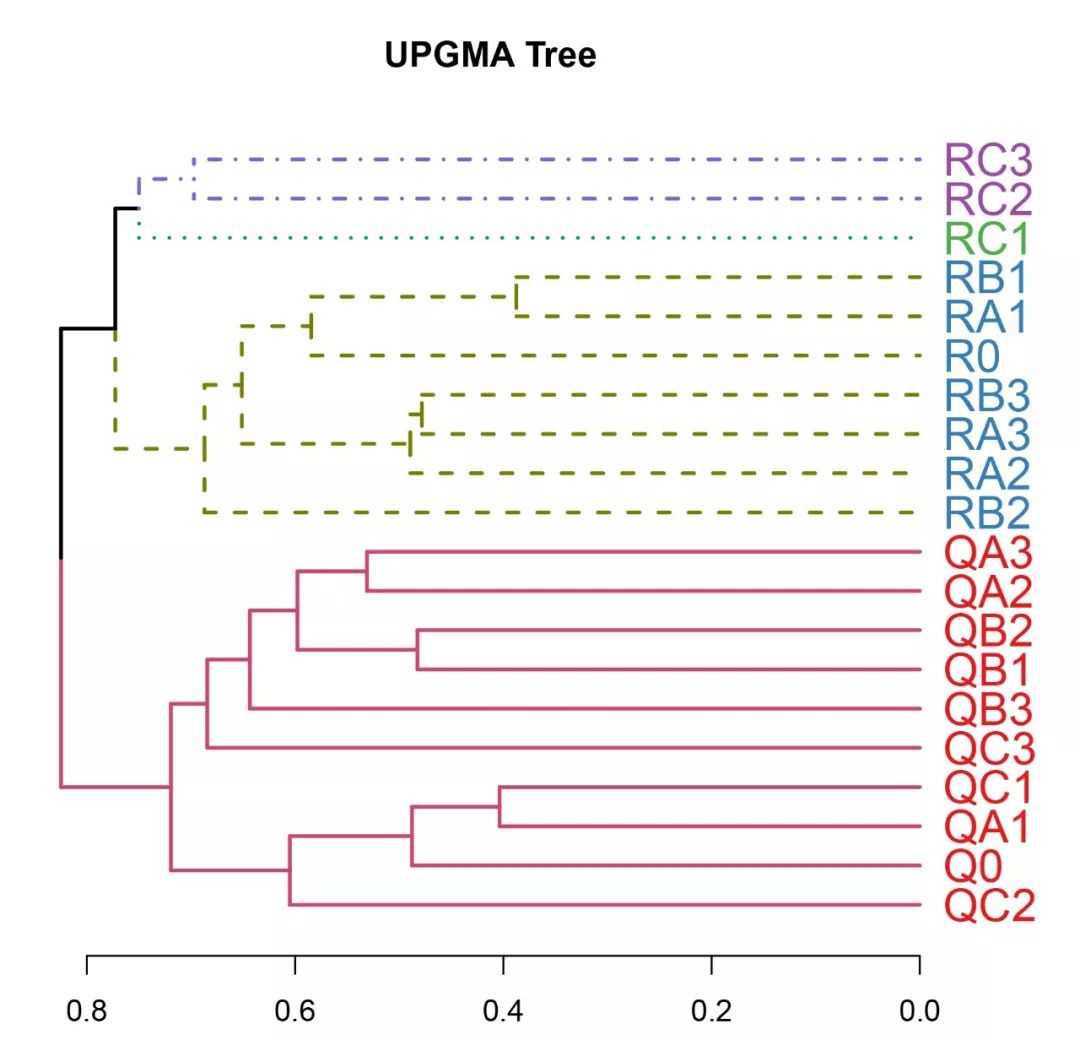
軟件包dendextend是一個很好的聚類樹可視化工具,可以使用“%>%”來對tree文件進行更新美化,如下所示:
hcd=hcd %>% set("labels_cex", 1.5) %>% set("branches_lwd", 2) %>% set("branches_k_color", k=4) %>% set("branches_k_lty", k=4) clusDendro=dendrapply(hcd, colLab)plot(clusDendro, main="UPGMA Tree", type="rectangle", horiz=TRUE)
感謝各位的閱讀,以上就是“R語言層次聚類與聚類樹怎么使用”的內容了,經過本文的學習后,相信大家對R語言層次聚類與聚類樹怎么使用這一問題有了更深刻的體會,具體使用情況還需要大家實踐驗證。這里是億速云,小編將為大家推送更多相關知識點的文章,歡迎關注!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。