您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章主要講解了“如何使用TCGAbiolinks分析TCGA中的表達譜數據”,文中的講解內容簡單清晰,易于學習與理解,下面請大家跟著小編的思路慢慢深入,一起來研究和學習“如何使用TCGAbiolinks分析TCGA中的表達譜數據”吧!
對于轉錄組數據而言,差異分析和富集分析是最核心的分析內容之一,通過TCGAbiolinks可以輕松實現TCGA表達譜數據的下載,差異分析,富集分析等功能,以乳腺癌的基因表達譜為例,分析過程如下
由于TCGA中乳腺癌的樣本很多,所以只挑選了部分樣本來測試,下載的過程如下
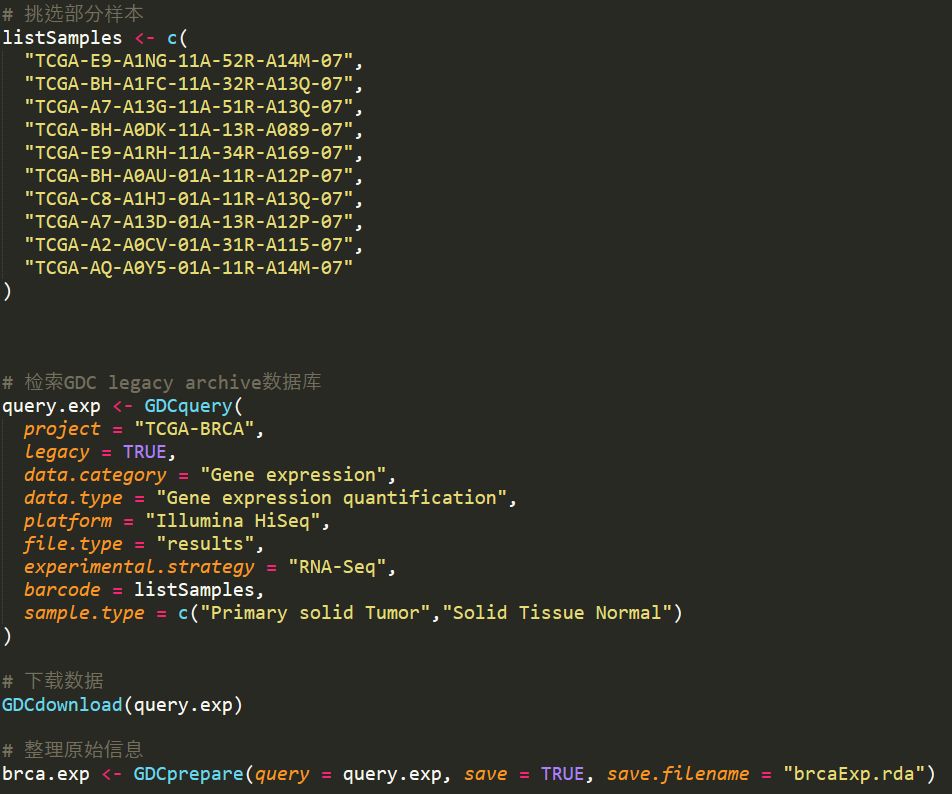
詳細步驟如下
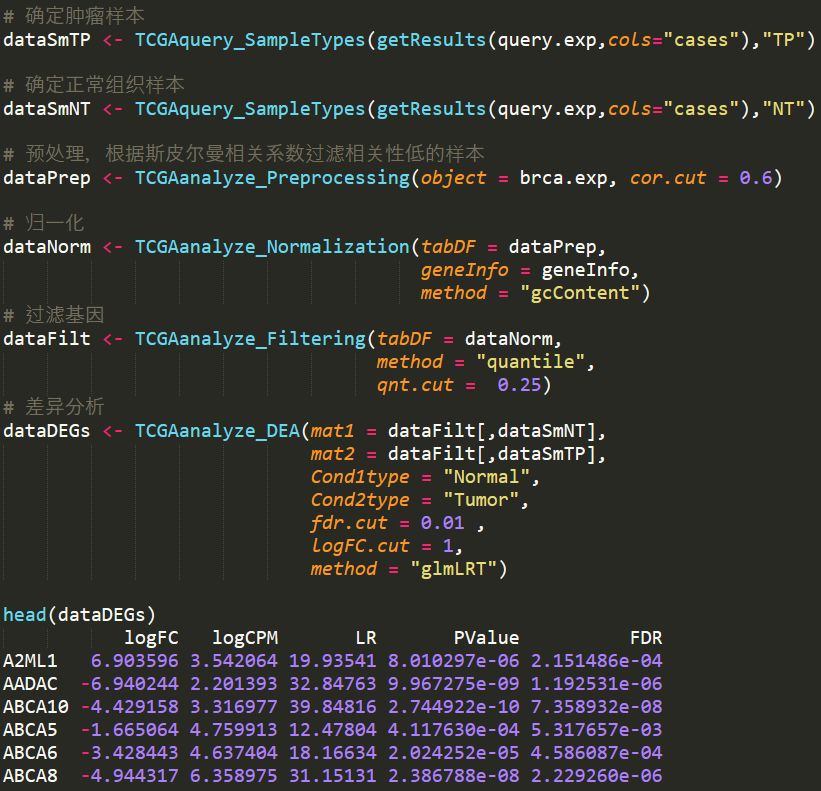
對數據進行預處理,根據樣本間的斯皮爾曼相關系數去除相關性較低的樣本
歸一化,調用了EDASeq中的歸一化算法
篩選基因,根據表達量的均值進行篩選
差異分析,調用了edgeR中的差異算法
代碼如下
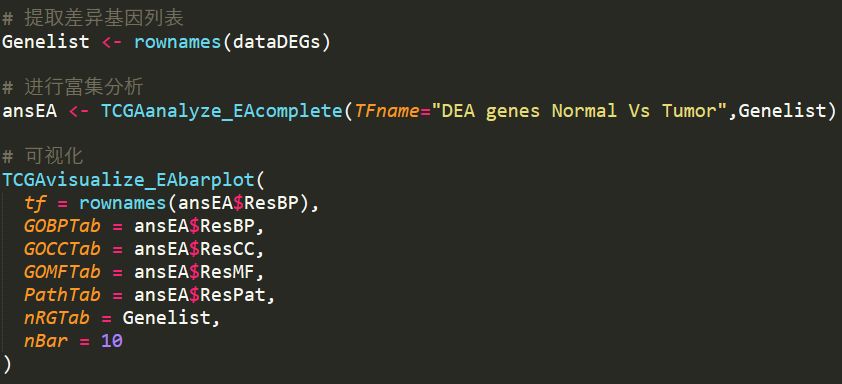
代碼如下
可視化的結果如下所示
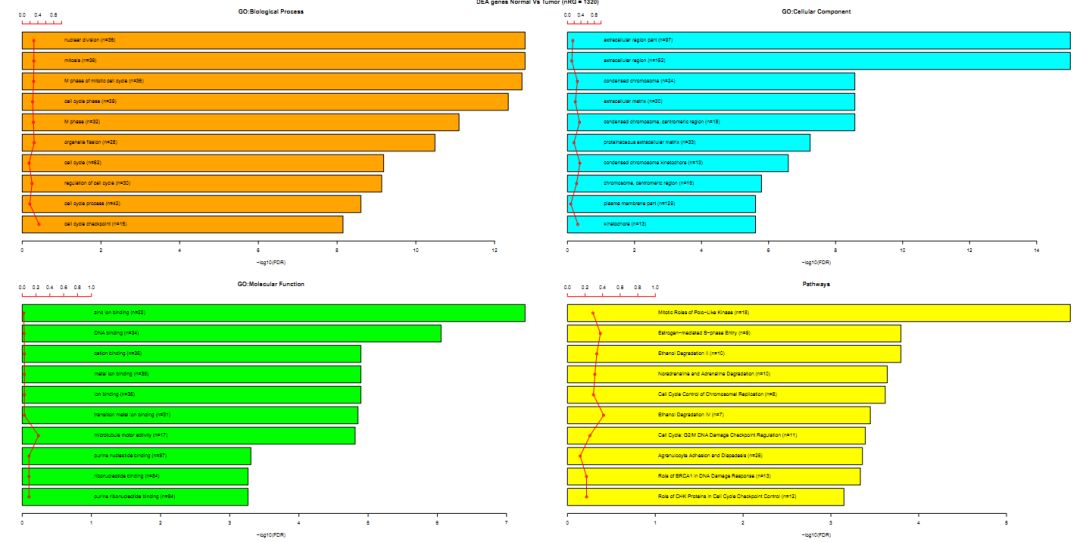
GO的3大類別加上kegg pathway共4個類別的數據,對應4張柱狀圖,每個柱狀圖展示的是FDR值最顯著的top10個條目,橫坐標我-log10(FDR), 散點代表的是GeneRatio, 也稱之為enrich factor, 代表富集到該通路下的差異基因個數占該通路下所有基因總數的比例。
感謝各位的閱讀,以上就是“如何使用TCGAbiolinks分析TCGA中的表達譜數據”的內容了,經過本文的學習后,相信大家對如何使用TCGAbiolinks分析TCGA中的表達譜數據這一問題有了更深刻的體會,具體使用情況還需要大家實踐驗證。這里是億速云,小編將為大家推送更多相關知識點的文章,歡迎關注!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。