您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章主要為大家展示了“HLAreporter軟件有什么用”,內容簡而易懂,條理清晰,希望能夠幫助大家解決疑惑,下面讓小編帶領大家一起研究并學習一下“HLAreporter軟件有什么用”這篇文章吧。
在前面的文章中,我們詳細介紹了HLA Allel的命名格式,示意圖如下
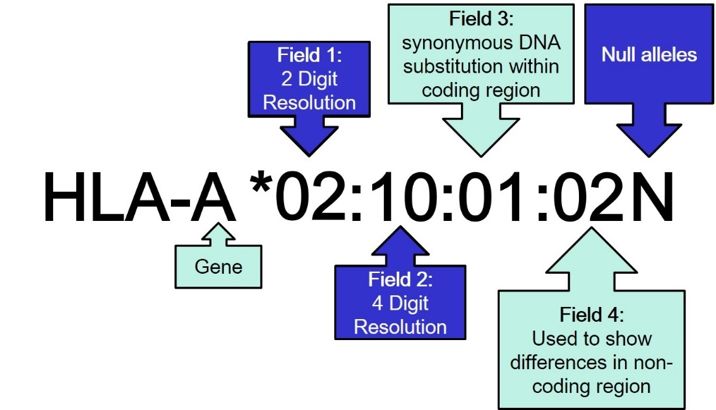
從示意圖可以看出,一個HLA Allel 可以分成四個字段,在加上最后的修飾后綴,共5個字段;在定義HLA 分型結果的分辨率時,會根據分型結果的最大位數來判斷,如果只給出了字段一,即血清學分類的信息,代表是2位的分型結果;如果最多給出了字段二,即對應的蛋白信息,代表是4位的分型結果;如果最多給出了字段三,即CDS區信息,代表是8位的分型結果;如果分型結果給出了最后的后綴,代表是9位的分型結果。
HLA分型的技術手段很多,比如芯片,高通量測序等;不同手段識別到的HLA Allel 分辨率不同,如果只能給出2位的分型結果,則屬于低分辨率;如果給出4位分型結果,屬于中分辨率;能夠給出8位或以上分型結果,屬于高分辨率。
本篇文章主要介紹HLA reporter 軟件,該軟件可以利用高通量測序的結果進行HLA 分型。其分型結果分辨率高,最多可到9位。下圖是該軟件與其他同類軟件的比較結果
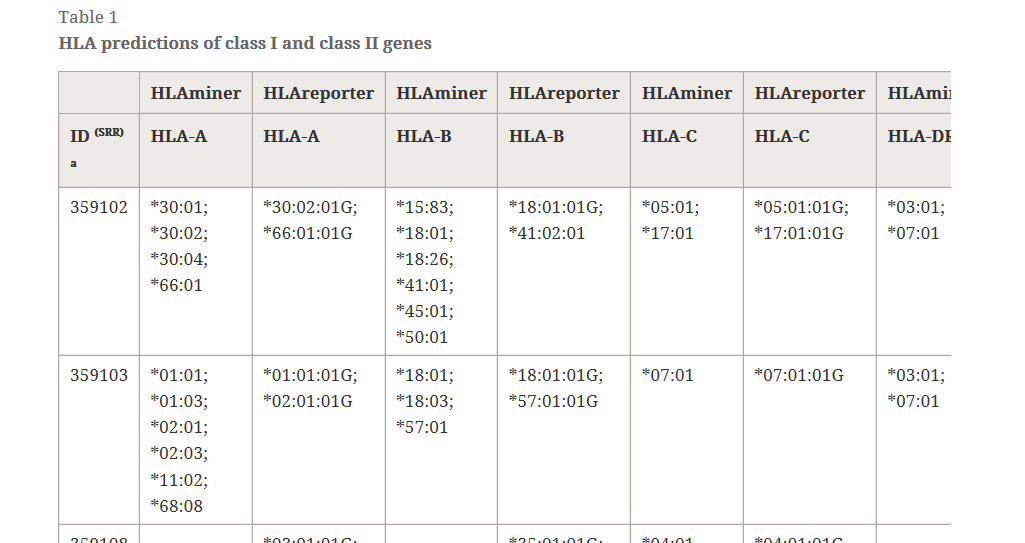
可以看到,在測試樣本中,HLAreporter 軟件分型結果由于HLAminer 的結果。該截圖來自于官方文獻,鏈接如下
https://genomemedicine.biomedcentral.com/articles/10.1186/s13073-015-0145-3
軟件的官網如下
http://paed.hku.hk/genome/software.html
安裝過程如下
wget http://paed.hku.hk/genome/software/HLAreporter.zip unzip HLAreporter.zip cd HLAreporter.v103
HLAreporter.sh就是該軟件的運行腳本。其原理如下
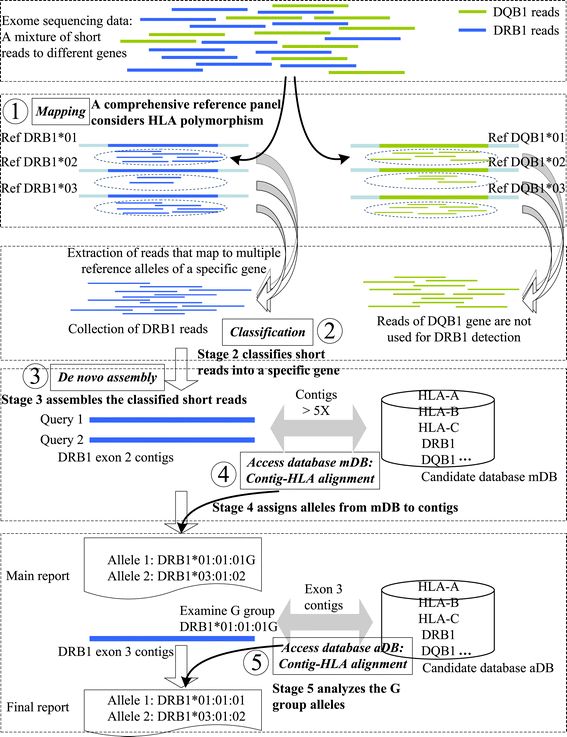
由于HLA的多態性,直接比對reads到參考基因組是不行的 ,HLAreporter 設計了一個CRP(comprehensive reference panel)參考基因組 , CRP 構建時參考了IMGT/HLA 數據庫中已知的HLA Allel信息,通過bwa將reads與CRP比對,提取比對到某個HLA基因的reads,然后進行組裝,將組裝的contig與數據庫比較,確定最終的Allel。
該軟件的用法如下
bash HLAreporter.sh test HLA_B test.R1.fq test.R2.fq
第一個參數test代表樣本名稱;第二個參數代表檢測的HLA基因,第三個和第四個參數代表雙端測序的fastq序列。
整個pipeline 分成了3個部分
當你提供了原始的fastq 數據時,會自動調用4digit_map_HLA.sh腳本進行比對
該腳本核心內容如下
bwa aln exon23_high_resolution_multi_ref.fa $1 > $3_1_exon23_high_resolution_multi_ref.sai bwa aln exon23_high_resolution_multi_ref.fa $2 > $3_2_exon23_high_resolution_multi_ref.sai bwa sampe exon23_high_resolution_multi_ref.fa $3_1_exon23_high_resolution_multi_ref.sai $3_2_exon23_high_resolution_multi_ref.sai $1 $2 > $3_exon23_high_resolution_multi_ref.sam samtools view -bS $3_exon23_high_resolution_multi_ref.sam > $3_exon23_high_resolution_multi_ref.bam samtools view -b -F 4 $3_exon23_high_resolution_multi_ref.bam > $3_exon23_high_resolution_multi_ref_mappedreads.bam samtools sort $3_exon23_high_resolution_multi_ref_mappedreads.bam $3_exon23_high_resolution_multi_ref_mappedreads_sorted samtools index $3_exon23_high_resolution_multi_ref_mappedreads_sorted.bam
$1和$2分別對應雙端測序的R1和R2端reads, $3表示樣本名稱,通過調用bwa sampe,將原始的雙端reads與exon23_high_resolution_multi_ref.fa比對,生成exon23_high_resolution_multi_ref_mappedreads_sorted.bam 文件。
對于HLA-A , HLA-B, HLA-C 這三個I型基因而言,調用4digit_predict_classI_ssake.sh腳本;對于HLA II 型基因,調用4digit_predict_classII_main.sh腳本。
利用ANFD數據庫中的信息,給出每個Allel在不同人群中的頻率,通過腳本HLAfreq.sh實現。
最終輸出結果保存在results目錄下,示意如下
------------------------------------------------------------------ HLAreporter version 1.03 Report created Fri Apr 17 11:24:24 HKT 2015 ------------------------------------------------------------------ Allele DRB1*01:01:01G Allele DRB1*04:07:01G Phase DRB1*04:08:01 Alleles detected DRB1*01:01:01G DRB1*01:17 DRB1*04:08:01 DRB1*04:07:01G Gap location: 97 208 Non-polymorphic gap: 111 bp HLA data quality profile: 10xcov% 100 20xcov% 100 30xcov% 97 50xcov% 89 ------------------------------------------------------------------ HLA allele frequency Four populations in Europe China Japan Africa are shown By allele frequency net database (www.allelefrequencies.net) ------------------------------------------------------------------ [Allele] [EUR] [CHN] [JPN] [AFR] DRB1*01:01 0.0860 0.0230 0.0582 0.0130 DRB1*04:07 0.0112 0.0030 0.0057 0.0030 DRB1*04:08 0.0039 0.0020 0.0000 0.0000 DRB1*01:17 0.0000 0.0000 0.0000 0.0000
HLAreporter目前只支持對以下11個基因的分型
HLA_A
HLA_B
HLA_C
HLA_DRB1
HLA_DRB2
HLA_DRB3
HLA_DRB4
HLA_DRB5
HLA_DQB1
HLA_DPB1
HLA_DQA1
以上是“HLAreporter軟件有什么用”這篇文章的所有內容,感謝各位的閱讀!相信大家都有了一定的了解,希望分享的內容對大家有所幫助,如果還想學習更多知識,歡迎關注億速云行業資訊頻道!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。