您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
本篇文章給大家分享的是有關怎樣使用R語言利用vcf格式文件計算核苷酸多樣性,小編覺得挺實用的,因此分享給大家學習,希望大家閱讀完這篇文章后可以有所收獲,話不多說,跟著小編一起來看看吧。
bcftools view snp.vcf.gz scaffold_1 > popgenome-vcf/scaffold_1
bcftools view snp.vcf.gz scaffold_2 > popgenome-vcf/scaffold_2
如果當前目錄下只有vcf格式文件,會遇到報錯Failed to open .vcf.gz: could not load index,可以參考 https://www.cnblogs.com/chenwenyan/p/11945445.html
tabix -p vcf snp.vcf.gz
如果當前目錄下沒有popgenome-vcf這個目錄,還需要新建目錄
mkdir popgenome-vcf
今天參考的文章里寫道 In theory, the r PopGenome can read VCF files directly, using the readVCF function. However, because our samples are haploid, we need to use a different function, r readData, which requires a folder with a separate VCF for each scaffold. 這個是為什么呢?
#install.packages("PopGenome")
library(PopGenome)
getwd()
setwd("VCF/")
snp<-readData("popgenome-vcf",format = "VCF")
get.sum.data(snp)
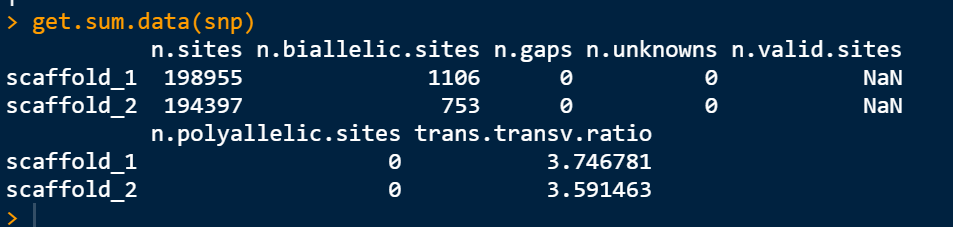
這里可以直接統計 轉換和顛換的比例
pops<-get.individuals(snp)[[1]]
pop1<-pops[grep("B\\.bam",pops)]
pop2<-pops[grep("b\\.bam",pops)]
pop1
pop2
snp<-set.populations(snp,list(pop1,pop2))
snp@populations
snp<-F_ST.stats(snp)
get.F_ST(snp)
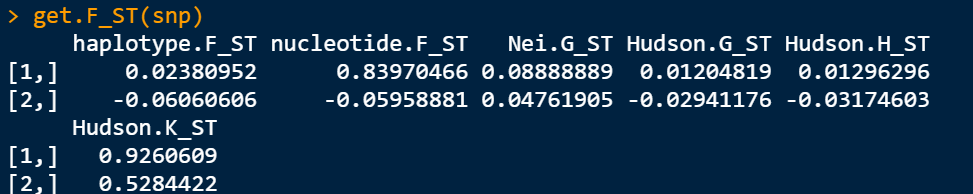
這里的指標都是什么意思呢?
get.diversity(snp)[[1]]
 這里的指標也看不懂是什么意思呀
這里的指標也看不懂是什么意思呀
win_snp<-sliding.window.transform(snp,
width = 10000,
jump = 2000,type = 2)
win_snp<-F_ST.stats(win_snp)
win_snp@nucleotide.F_ST
win_snp@nuc.diversity.within
library(ggplot2)
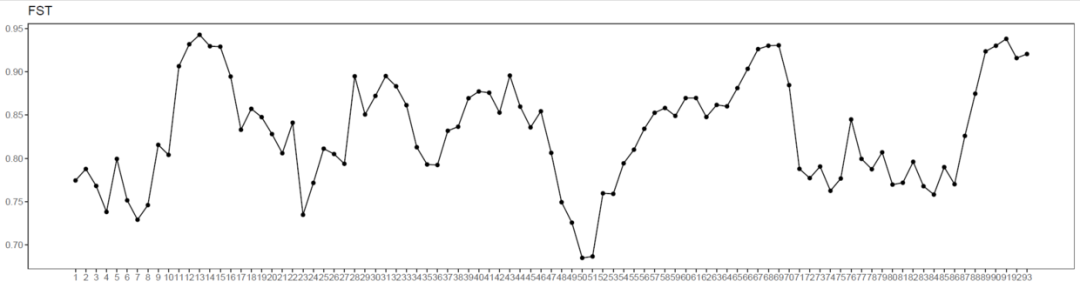
win_fst <- data.frame(x=1:dim(win_snp@nucleotide.F_ST)[1],
y=win_snp@nucleotide.F_ST[,1])
head(win_fst)
p1<-ggplot(win_fst,aes(x=x,y=y))+
geom_point()+
geom_line()+
theme_bw()+
theme(panel.grid = element_blank())+
scale_x_continuous(breaks = win_fst$x,
labels = win_fst$x)+
labs(x=NULL,y=NULL,title = "FST")
ggsave("FST.pdf",p1,width = 15,height = 4)
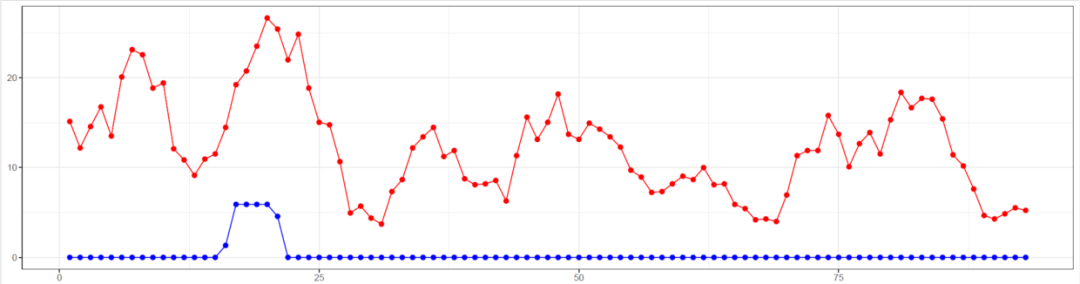
bb_div <- win_snp@nuc.diversity.within[,1] # diversity among B (bb = "big B")
lb_div <- win_snp@nuc.diversity.within[,2] # diversity among B (lb = "little b")
bb_div
df1<-data.frame(x=1:length(bb_div),y=bb_div)
df2<-data.frame(x=1:length(lb_div),y=lb_div)
p2<-ggplot()+
geom_line(data=df1,aes(x=x,y=y),color="red")+
geom_point(data=df1,aes(x=x,y=y),size=2,color="red")+
geom_line(data=df2,aes(x=x,y=y),color="blue")+
geom_point(data=df2,aes(x=x,y=y),size=2,color="blue")+
theme_bw()+labs(x=NULL,y=NULL)
ggsave("diversity.pdf",p2,width = 15,height = 4)
以上就是怎樣使用R語言利用vcf格式文件計算核苷酸多樣性,小編相信有部分知識點可能是我們日常工作會見到或用到的。希望你能通過這篇文章學到更多知識。更多詳情敬請關注億速云行業資訊頻道。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。